2. 青岛大学附属医院消化内科,山东 青岛 266071;
3. 青岛大学医学研究中心,山东 青岛 266071
遗传性胰腺炎(HP) 是一种罕见的常染色体显性遗传性疾病,约占胰腺炎总数的1%[1],可以引发急性复发性和慢性胰腺炎[2]。目前已知的HP相关基因主要有人阳离子胰蛋白酶原(PRSS1)、丝氨酸肽酶抑制因子Kazal型1(SPINK1)、糜蛋白酶C(CTRC)、囊性纤维跨膜转导调节子(CFTC)和羧肽酶A1(CPA1)[3]。其中,PRSS1基因突变被认为是导致HP的主要原因。我们在前期研究中发现1例不明原因的反复发作的急性胰腺炎病人携带PRSS1 c.455-33C>T内含子杂合突变[4],在clinvar中未发现这一突变,在PubMed中未见相关突变的病例报道。内含子突变可影响选择性剪切,可作为转录增强子影响基因的表达[5]。本文对该临床表型明确病人所携带的新突变PRSS1 c.455-33C>T进行功能研究以明确其致病性。
1 资料和方法 1.1 临床资料病人,女,69岁。因“间断上腹痛”于2020年6月首次发病入院。病人无明显诱因出现持续性上腹痛,无肩部及背部放射痛,弯腰抱膝位可稍缓解。既往无吸烟及饮酒史,无外伤手术史,否认家族遗传病史。入院体检:上腹肌紧张,有压痛、无反跳痛,其余未见异常。实验室检查:白细胞增多(12.60×109/L),中性粒细胞增多(9.98×109/L),血淀粉酶明显增高(1 953.00 U/L),血糖明显升高(17.6 mmol/L)。上腹部核磁动态增强扫描示胰头稍饱满、主胰管略扩张,胰胆管核磁造影成像示主胰管略扩张。诊断为急性胰腺炎。入院给予禁饮食、抑酸、生长抑素抑制胰液分泌、乌司他丁抑制胰酶活性及补液等治疗,病人病情好转出院。其后病人类似症状反复发作数次,均住院经非手术治疗后症状缓解出院。为了进一步明确诊断,对病人进行了PRSS1基因热点突变检测,发现PRSS1 c.455-33C>T内含子杂合突变。
1.2 网站突变功能预测在网站clinvar(https://www.ncbi.nlm.nih.gov/clinvar/?gr=0&term=prss1%5Bgene%5D&redir=gene)中对PRSS1 c.455-33C>T突变进行查找,确定是否收录该突变;然后在Lncipedia网站中(https://lncipedia.org/)对该突变进行了lncRNA预测;在PubMed(https://pubmed.ncbi.nlm.nih.gov/)中对该突变进行检索。
1.3 PRSS1 c.455-33C>T突变剪切功能及增强子功能验证 1.3.1 实验材料人胚肾Hek293T细胞(武汉普诺赛生命科技有限公司);Lipofectamin3000转染试剂盒-L3000001、FastDigest enzyme-FD0524(Thermo Fisher Scientific公司);DMEM培养基(meilunbio,MA0212);RNA-easy Isolation Reagent-R701、2×Phanta® Flash Master Mix-P510-01、2×Clon Express Mix-C115-01AA、2×Universal Ligation Mix-C311-01/02、FastPure EndoFree Plasmid Maxi Kit (DC202,Vazyme);Evo M-MLV反转录试剂盒(Accurate Biology公司,AG11711);柱式血液基因组DNA抽提试剂盒-B518253-0100,Agarose以及Regular (生工生物工程公司,A620014);50×TAE缓冲液-T1060、100 bp DNA Ladder-M1200、6×DNA Loading-D1010,LB营养琼脂-L8290,LB肉汤-L8291(Solarbio);TS-GelRed核酸染料-TSJ003,Tstbl3感受态细胞,pGL4.23质粒,包含PRSS1 intron3所有序列的质粒,DNA凝胶回收试剂盒-TSP601(北京擎科生物科技有限公司);pSPL3质粒(赠与);无内毒素质粒小提试剂盒(康为世纪公司,CW2106S);Dual-Luciferase® Reporter Assay System(Promega,E1960);引物及其序列(华大基因)见表 1。
| 表 1 引物名称及其序列 |
|
|
将Hek293T细胞复苏后放入细胞培养箱中进行培养,待细胞培养到一定数量后消化接种于35 mm细胞培养皿,按照Lipofectamin3000转染试剂盒说明书进行质粒转染。
1.3.3 质粒构建采集健康对照者外周血3mL于EDTA抗凝管中,应用血液基因组DNA抽提试剂盒提取DNA,利用表 1中引物3、4将PRSS1基因intron3序列和PRSS1基因intron3-exon4-intron4序列扩增后,分别与pGL4.23、pSPL3载体(表 1中1、2)连接(图 1),转化Tstbl3感受态细胞,在LB固体培养基上37 ℃过夜培养,挑取单菌落进行测序(华大基因),取测序成功菌液扩大培养后进行质粒小提。将质粒分别命名为EWT、SWT。
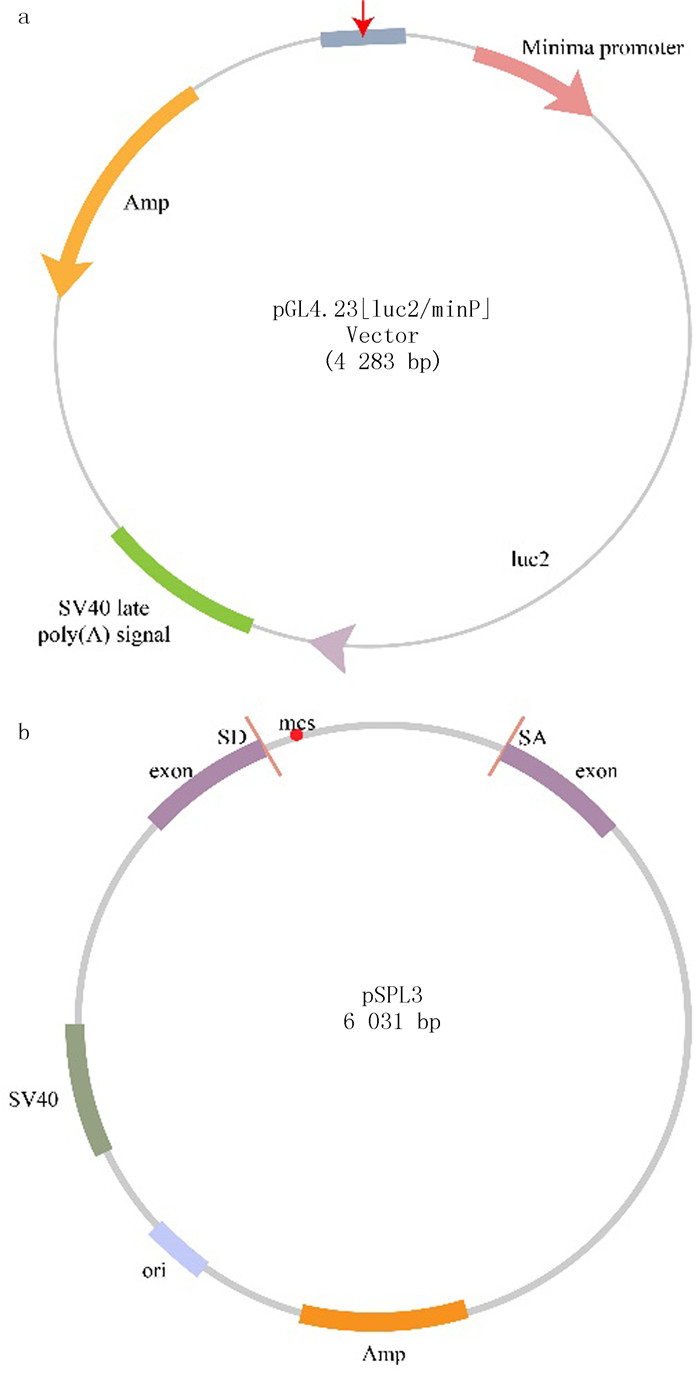 |
| a:以pGL4.23为载体插入PRSS1 intron3基因序列构建EWT质粒;b:以pSPL3为载体插入PRSS1 intron3-exon4-intron4基因序列构建SWT质粒。 图 1 质粒构建示意图 |
在EWT、SWT的基础上用表 1中定点突变引物5进行扩增后,用2×Universal Ligation Mix进行连接,转化Tstbl3感受态细胞,在LB固体培养基上37 ℃过夜培养,挑取单菌落进行测序(华大基因),将测序成功菌液扩大培养后进行质粒小提,将质粒分别命名为E33、S33。
1.3.5 逆转录聚合酶链式反应(RT-PCR)扩增目的片段① 扩增目的基因片段及点突变扩增:总反应体系50 μL,内含2×Phanta® Flash Master Mix 25 μL、上下游引物各2 μL、DNA 1 μL、超纯水20 μL。置于PE9700热循环仪上,程序为:98 ℃ 30 s;98 ℃ 10 s,60 ℃ 5 s,72 ℃ 5 s,循环35次;72 ℃ 1 min。②RT-PCR:总反应体系10 μL,内含DNA聚合酶5.0 μL、上下游引物各0.5 μL、DNA 1.0 μL、超纯水3.0 μL。置于PE9700热循环仪上,程序为:94 ℃ 5 min;98 ℃ 30 s;65 ℃ 30 s,0.5 ℃/Cycle,72 ℃ 1 min循环20次,94 ℃ 30 s,55 ℃ 30 s,72 ℃ 1 min,循环15次,72 ℃延长5 min。最后4 ℃保存。③逆转录:反应体系及程序均按照Evo M-MLV反转录试剂盒说明书进行。
1.3.6 凝胶电泳检测扩增片段将0.45 g琼脂糖粉末加入30 mL的1×TAE溶液中配制15 g/L制胶液,并加入3 μL的10 000×GelRed后倒入制胶器,电源设置120 V,进行20 min凝胶电泳。
1.3.7 荧光素酶活性的检测将质粒EWT、E33 500 ng分别与Renilla Luciferase内参质粒2.5 ng共转入24孔板中的Hek293T细胞,转染48 h后弃培养液,每孔加入100 μL 1×Passive Lysis Buffer,于摇床上室温摇15 min。按照双荧光素酶报告基因分析系统(Promega)操作手册进行荧光素酶活性测定。
1.4 统计学处理使用GraphPad Prism 9.0软件进行统计学处理。计量资料比较采用t检验,P < 0.05表示差异有统计学意义。
2 结果 2.1 突变初步分析在clinvar网站中对PRSS1 c.455-33C>T突变进行查找发现并没有收录该突变;对该突变进行lncRNA预测发现,该区域没有已知的lncRNA;检索PubMed也没有对该突变的报道。
2.2 PRSS1 c.455-33C>T突变剪切功能验证为验证PRSS1 c.455-33C>T突变是否改变基因的剪切,将构建好的SWT、S33质粒以及pSPL3空载质粒转染Hek293T细胞,对转染后的细胞RNA进行RT-PCR检测该突变引入pSPL3载体后的mRNA剪切是否发生改变。pSPL3的两个外显子分别为92 bp和171 bp,插入的PRSS1 exon4大小为137 bp(图 2a),SWT质粒预测有2种剪切结果,见图 2b。对RT-PCR产物进行凝胶电泳显示,SWT与S33组产物相同,分别为400 bp和263 bp(图 2c),符合图 2b中的预测结果。表明PRSS1 c.455-33C>T突变并不能引起剪切功能的改变。
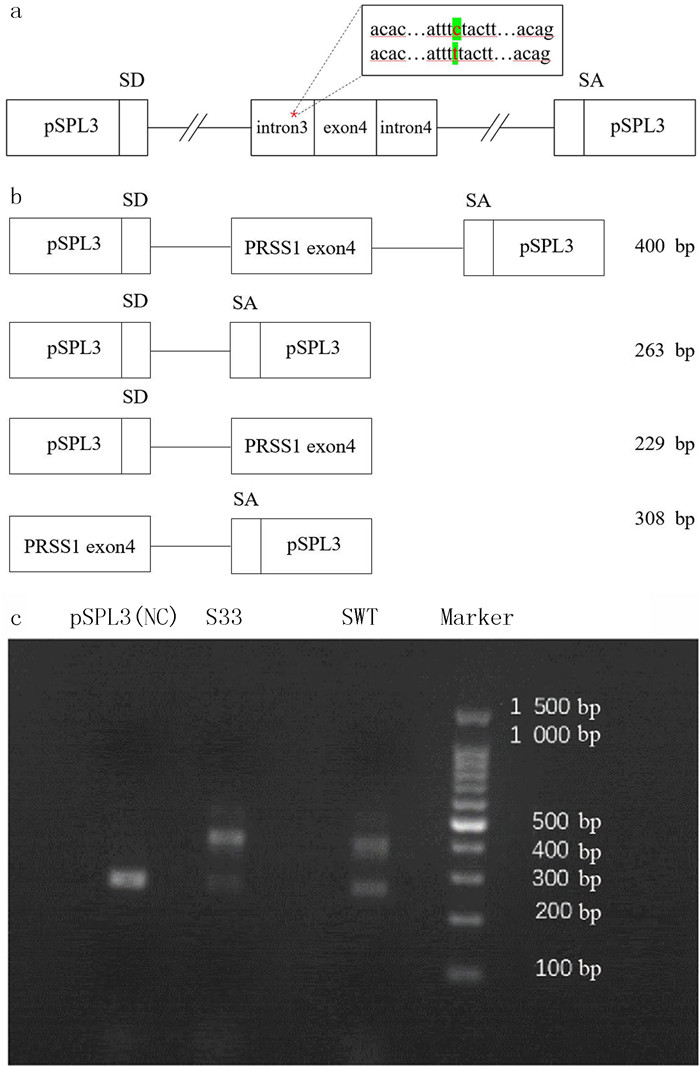 |
| a:将分析序列克隆到pSPL3载体中,SA为接头受体部位,SD为剪接供体部位;b:剪切结果预测;c:PRSS1 c.455-33C>T变体产生了与正常基因相对应的400 bp以及263 bp的产物。 图 2 RT-PCR法检测PRSS1 c.455-33C>T突变对剪切功能的影响 |
pGL4.23载体含Luciferase荧光素酶报告基因及Minimal promoter基因,用于研究目的基因的增强子功能,若Luciferase荧光素酶活性上升,说明该段Minimal promoter受到增强子的调控。本研究构建以pGL4.23为载体的EWT、E33质粒,转染Hek293T细胞后通过双荧光素酶报告基因实验来验证c.455-33C>T突变是否有增强子功能。结果示EWT组Ratio值高于E33组(n=7,t=12.23,P < 0.001),见图 3。提示PRSS1 c.455-33C>T突变并没有增强子功能。
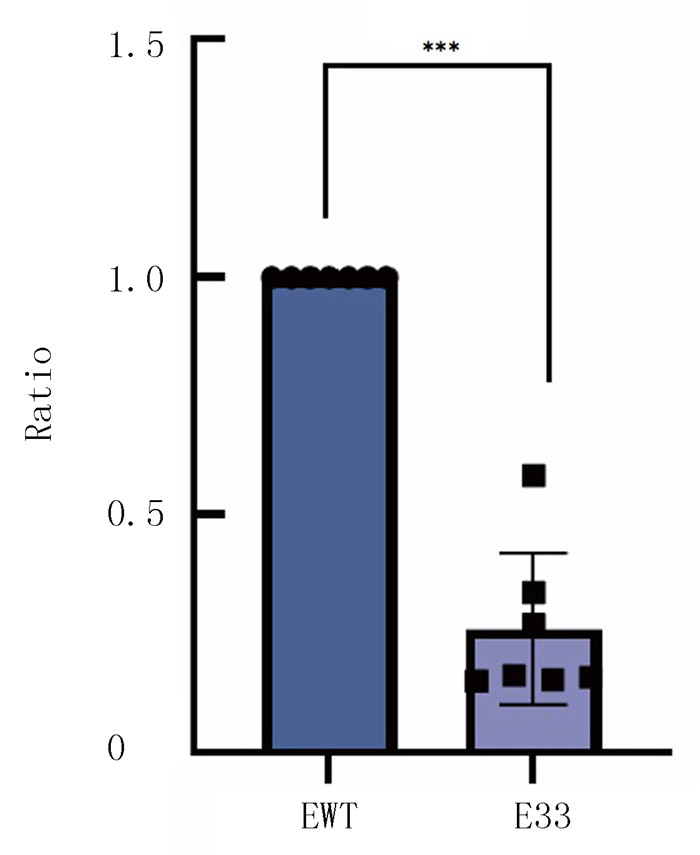 |
| ***P<0.001。 图 3 EWT、E33两组Ratio值比较 |
在临床中,当病人满足以下1个或多个标准时应考虑HP,并行基因检测[6]:①特发性慢性胰腺炎(CP)、复发性急性胰腺炎(RAP)家族史或儿童时期不明原因胰腺炎;②有与HP相关的已知突变的亲属;③年龄25岁以下且患特发性CP;④原因不明的RAP。HP临床表现多样、无特异性,包括腹痛、恶心呕吐、食欲减退、餐后上腹饱胀、脂肪泻或腹泻、营养吸收障碍、内外分泌功能受损、腹腔积液、脾大、胆管和十二指肠梗阻、阻塞性黄疸、假性囊肿、胰腺癌等,发作时血尿淀粉酶、脂肪酶等升高,这与其他原因引起的胰腺炎表现相类似[7-9]。1996年WHITCOMB等[10]和REBOURS等[11]在HP病人中发现第7号染色体长臂(7q35)上的PRSS1基因发生突变,与HP发病相关。PRSS1基因突变通过减少胰凝乳蛋白酶(CTRC)依赖性胰蛋白酶原降解、增加CTRC介导的激活肽加工或者直接刺激自身激活而增强了PRSS1的激活,从而导致胰腺炎发生[12]。对PRSS1基因的深入研究可以为HP的发病机制提供新的线索[13-15]。
PRSS1突变HP病人的影像学表现包括:胰腺萎缩、钙化和胰腺主导管扩张,称为PRSS1成像三联征[16]。本例胰腺炎病人影像学提示主胰管扩张,且多次因RAP入院治疗,排除其他常见RAP诱因外应注意存在HP的可能。本文对该病人行基因检测,发现该病人存在PRSS1 c.455-33C>T突变,且该内含子突变目前尚未有报道。
多年来,基因组序列主要侧重于与其编码蛋白质能力有关的外显子,认为内含子序列在很大程度上是无功能的。然而,最近研究支持内含子也有功能,例如一些启动子与位于内含子内的调节序列结合发挥转录增强子的作用[17];一些内含子并不会被完全降解,产生功能性非编码RNA副产物发挥作用[18];也有一些内含子可作为“滞留”内含子来暂时保留在核转录本中,但仍可剪接,也可以通过其他机制影响基因表达;还有一些内含子通过引起非常规剪切来起作用。近年来随着全基因组测序的出现,发现很多遗传性疾病是由内含子的突变引起的。本文通过构建能够用于剪切分析的SWT、S33质粒,将SWT、S33质粒分别转染Hek293T细胞后进行分析,结果显示PRSS1 c.455-33C>T突变并不引起剪切的改变,表明该突变不是通过影响剪切导致HP的。之后又以pGL4.23为载体构建了可用于检测插入片段的增强子活性的EWT、E33质粒,双荧光素酶实验结果显示插入的突变片段导致启动子功能减弱。然而PRSS1基因突变存在功能获得性突变时才会引起HP,所以PRSS1 c.455-33C>T突变不会直接导致PRSS1的表达增强,但是这一突变具体影响哪个基因的启动子,现在还不清楚;该突变是否通过抑制某个基因的转录而促进胰腺炎发生的可能性也不能排除。后续也可以在这方面进行深入的研究。
综上所述,随着对RAP和慢性胰腺炎病人进行常规遗传筛查,已知易感基因中罕见或者个体变异的发现已变得越发普遍。在缺乏遗传证据的情况下,对变异体的功能分析可能会揭示其临床意义[13]。因此,对于新发现的内含子突变位点进行剪切分析、探索增强子功能和非编码RNA的相关性,对于其致病性判断就显得尤为重要。
(感谢意大利罗马Sapienza大学的Bottillo Irene教授为本研究提供pSPL3载体!)
| [1] |
SINGHI A D, PAI R K, KANT J A, et al. The histopathology of PRSS1 hereditary pancreatitis[J]. American Journal of Surgical Pathology, 2014, 38(3): 346-353. DOI:10.1097/PAS.0000000000000164 |
| [2] |
WEISS F U. Pancreatic cancer risk in hereditary pancreatitis[J]. Frontiers in Physiology, 2014, 5: 70. |
| [3] |
MASAMUNE A. Genetics of pancreatitis: the 2014 update[J]. The Tohoku Journal of Experimental Medicine, 2014, 232(2): 69-77. DOI:10.1620/tjem.232.69 |
| [4] |
张文清, 雷珂, 魏宏云, 等. PRSS1和SPINK1基因热点突变检测对遗传性胰腺炎高危人群的诊断价值[J]. 精准医学杂志, 2022, 8(2): 157-160. |
| [5] |
VAZ-DRAGO R, CUSTÓDIO N, CARMO-FONSECA M. Deep intronic mutations and human disease[J]. Human Gene-tics, 2017, 136(9): 1093-1111. DOI:10.1007/s00439-017-1809-4 |
| [6] |
SHELTON C A, UMAPATHY C, STELLO K, et al. Here-ditary pancreatitis in the United States: survival and rates of pancreatic cancer[J]. American Journal of Gastroenterology, 2018, 113(9): 1376. DOI:10.1038/s41395-018-0194-5 |
| [7] |
KLEEFF J, WHITCOMB D C, SHIMOSEGAWA T, et al. Chronic pancreatitis[J]. Nature Reviews Disease Primers, 2017, 3: 17060. DOI:10.1038/nrdp.2017.60 |
| [8] |
RAMALHO G X, DYTZ M G. Diabetes of the exocrine pancreas related to hereditary pancreatitis, an update[J]. Current Diabetes Reports, 2020, 20(6): 16. DOI:10.1007/s11892-020-01299-8 |
| [9] |
SUZUKI M, MINOWA K, NAKANO S, et al. Genetic abnormalities in pancreatitis: an update on diagnosis, clinical features, and treatment[J]. Diagnostics, 2020, 11(1): 31. DOI:10.3390/diagnostics11010031 |
| [10] |
WHITCOMB D C, PRESTON R A, ASTON C E, et al. A gene for hereditary pancreatitis maps to chromosome 7q35[J]. Gastroenterology, 1996, 110(6): 1975-1980. DOI:10.1053/gast.1996.v110.pm8964426 |
| [11] |
REBOURS V, LÉVY P, RUSZNIEWSKI P. An overview of hereditary pancreatitis[J]. Digestive and Liver Disease: Official Journal of the Italian Society of Gastroenterology and the Italian Association for the Study of the Liver, 2012, 44(1): 8-15. |
| [12] |
MAYERLE J, SENDLER M, HEGYI E, et al. Genetics, cell biology, and pathophysiology of pancreatitis[J]. Gastroente-rology, 2019, 156(7): 1951-1968. DOI:10.1053/j.gastro.2018.11.081 |
| [13] |
JANCSÓ Z, ORACZ G, KUJKO A A, et al. Novel pathogenic PRSS1 variant p.Glu190Lys in a case of chronic pancreatitis[J]. Frontiers in Genetics, 2019, 10: 46. DOI:10.3389/fgene.2019.00046 |
| [14] |
GAO F. PRSS1_p.Leu81Met mutation results in autoimmune pancreatitis[J]. World Journal of Gastroenterology, 2013, 19(21): 3332. DOI:10.3748/wjg.v19.i21.3332 |
| [15] |
TEICH N, ROSENDAHL J, TÓTH M, et al. Mutations of human cationic trypsinogen (PRSS1) and chronic pancreatitis[J]. Human Mutation, 2006, 27(8): 721-730. DOI:10.1002/humu.20343 |
| [16] |
O'BRIEN C M, O'BRIEN A C, GLEESON L, et al. Imaging of genetically-mediated pancreatitis[J]. Clinical Imaging, 2020, 65: 113-118. |
| [17] |
YASUDA H, OH C D, CHEN D, et al. A novel regulatory mechanism of type Ⅱ collagen expression via a SOX9-depen-dent enhancer in intron 6[J]. Journal of Biological Chemistry, 2017, 292(2): 528-538. |
| [18] |
HUBÉ F, FRANCASTEL C. Mammalian introns: when the junk generates molecular diversity[J]. International Journal of Molecular Sciences, 2015, 16(3): 4429-4452. |
 2024, Vol. 60
2024, Vol. 60

