银屑病是一种免疫相关的以角化细胞异常增殖为主要特征的皮肤病,影响了全球约1.25亿人[1-2]。目前,银屑病的治疗已经从单纯的外用药物治疗转变为外用药物、系统用药、光疗和生物制剂等的联合治疗[3-4]。随着基因测序技术的广泛应用,与银屑病相关的基因已经逐渐被揭示,如CXCL10、CXCL8、STAT1、SPRR1B、ISG15等基因在银屑病病灶中存在显著的表达差异,并被预测为可能的基因指标[5-7]。治疗试验显示,布罗达单抗(brodalumab)治疗后病损组织S100A7A、KRT16等基因表达量发生显著变化[8-9]。依那西普(etanercept)治疗后白细胞介素1B(IL-1B)、白细胞介素19(IL-19)等炎症相关细胞因子的基因表达出现下调[10]。不同疗法对银屑病皮损组织基因表达的影响被逐步揭示,其可能的相互作用有待探究[5]。本研究收集了近年来银屑病临床治疗试验的基因表达数据,探究不同疗法对银屑病皮损组织基因表达的影响及其相互作用,以期为银屑病临床治疗提供相关参考。
1 资料和方法 1.1 数据检索从GEO(Gene Expression Omnibus)数据库(https://www.ncbi.nlm.nih.gov/geo/)中,以“acitretin OR adalimumab OR apremilast OR brodalumab OR calcipotriol OR certolizumab pegol OR ciclosporin OR corticosteroid OR etanercept OR fumarates OR glucocorticoids OR guselkumab OR infliximab OR ixekizumab OR methotrexate OR retinoic acid OR risankizumab OR secukinumab OR tildrakizumab OR tretinoin OR ustekinumab OR UVB AND psoriasis”为检索式,检索不同疗法治疗银屑病的数据集。纳入标准:①样本为人皮肤组织或角质形成细胞;②包含至少一种治疗方法干预的样本及相应的对照组。排除标准:①无应用单一疗法的干预组;②无对照组。以“psoriasis”为检索式从GEO数据库中检索银屑病病变组织基因表达数据集。纳入标准:样本为寻常型银屑病的皮损部位活检组织,包含非皮损部位的对照。
1.2 数据质控及预处理利用R软件对收集的数据进行质控,通过绘制灰度图、权重图、残差图及残差符号图对单个芯片数据进行初步质控。通过绘制相对对数表达(RLE)箱线图、相对标准差(NUSE)箱线图以及RNA降解图,对整组芯片数据进行质控,并剔除不合格样品。通过rma算法对芯片数据进行背景修正、系统校正及表达值综合。将同种干预措施的数据组合并。
1.3 差异基因(DEGs)筛选在R软件中通过limma包按照经验贝叶斯(Bayes)法对各基因的表达差异进行统计学检验,并以Benjamini and Hochberg(BH)法校正P值。以校正P值(adjusted P) < 0.05,且对数化的倍数变化(|log2FC|)≥1.5为标准筛选DEGs。
1.4 DEGs分析不同治疗组的DEGs与银屑病组取交集,各治疗组两两之间分别取交集。银屑病组和各治疗组中的上调、下调DEGs交叉匹配。将交集基因导入STRING数据库组建蛋白质-蛋白质相互作用(PPI)网络。通过Cytoscape软件的cytoHubba插件以及MCODE插件识别相互作用密切的基因簇。后续步骤中根据功能富集结果对基因簇进行重新评估和调整。通过GO和KEGG分析对基因簇的功能进行预测和分析,同时分析了未集中在基因簇中以及未匹配的DEGs的功能。
2 结果 2.1 数据集治疗组共收集了17个数据集包括608份样本,经质量控制后选取568个样本进行进一步分析。确定了12种不同的疗法。银屑病组共检索到6个数据集包括435份样本,其中412份样本可用于进一步分析。见表 1、图 1A。
| 表 1 基因数据集概况 |
|
|
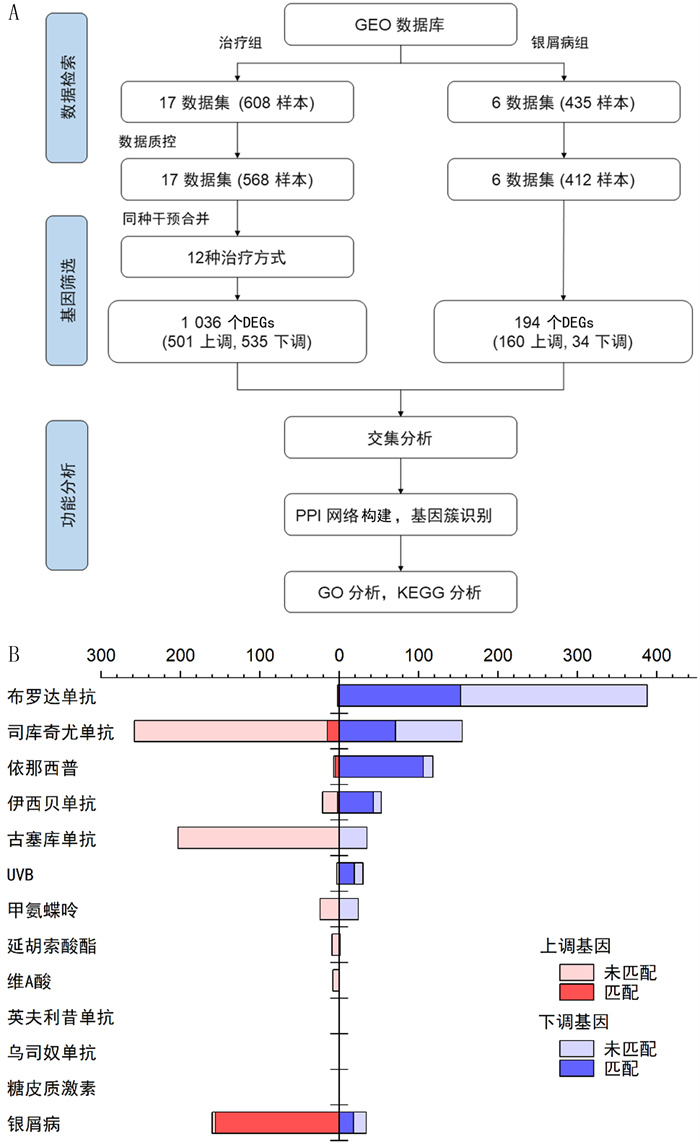 |
| A:研究流程图;B:DEGs数目。 图 1 研究流程图及DEGs |
治疗组中发现1 036个DEGs(501个上调,535个下调),银屑病组共检测到194个DEGs(160个上调,34个下调)。治疗组中共有156个下调基因与银屑病组中的上调基因匹配,同时有18个上调基因可与银屑病组中的下调基因匹配。见图 1B。
2.3 PPI网络和基因簇基于银屑病组与治疗组交集DEGs建立PPI网络,识别了6个关键基因簇。同簇内的基因(如簇2和4)有密切的内在联系,而某些簇(如簇3和5)则与其他基因间有广泛的联系(图 2)。治疗组中,多数单克隆抗体治疗组的DEGs与关键基因簇之间存在广泛的联系,如依那西普和布罗达单抗,而延胡索酸酯(fumarates)、甲氨蝶呤(methotrexate)等治疗组的DEGs与关键基因簇的相关性较弱。
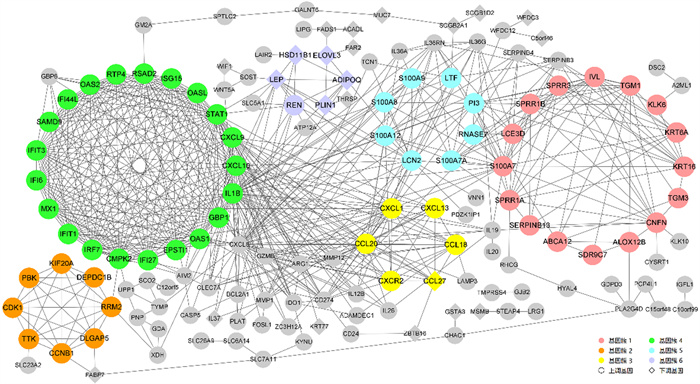 |
| 图 2 交集DEGs的PPI网络 |
通过GO和KEGG的富集分析结果对关键基因簇的功能进行推测,结果显示,基因簇1与角质细胞分化(GO: 0030216)和表皮发育(GO: 0008544)有关;基因簇2的基因与有丝分裂(GO: 1901992)以及细胞周期(GO: 0090068, hsa04110)密切相关;基因簇3则主要与趋化作用(GO: 0030595)和趋化因子(GO: 0008009, hsa04062)相关;簇4和簇5的基因分别与防御病毒(GO: 0051607)和防御细菌、真菌(GO: 0042742, GO: 0050832)相关;而基因簇6则主要与分解代谢的过程有关(GO: 0016042)。此外,本研究还发现基因簇1、4和5均与IL-17信号通路相关(hsa04657)。
治疗组的未匹配基因的功能分析结果显示,布罗达单抗治疗组中的下调基因与有丝分裂(GO: 0140014)和细胞周期(hsa04110)相关,司库奇尤单抗(secukinumab)和UVB组中均发现了与角化相关的基因(GO: 0030216);同时,古塞库单抗(guselkumab)组的上调基因和伊西贝单抗(ixekizumab)、司库奇尤单抗组的下调基因中发现了IL-17通路相关的基因(hsa04657)。见图 3、4。
 |
| A~F展示了基因簇1~6中的基因与主要功能集的联系。BP:生物过程;CC:细胞成分;MF:分子功能。 图 3 基因簇的GO富集结果 |
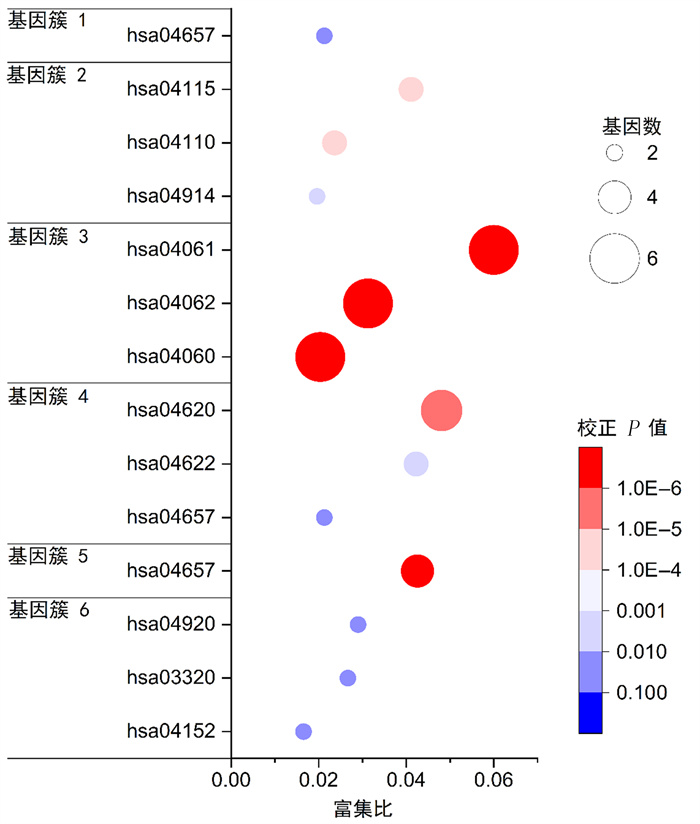 |
| hsa04657:IL-17信号通路;hsa04115:p53信号通路;hsa04110:细胞周期;hsa04914:孕激素介导的卵母细胞成熟;hsa04061:病毒蛋白与细胞因子及细胞因子受体相互作用;hsa04062:趋化因子信号通路;hsa04060:细胞因子-细胞因子受体相互作用;hsa04620:toll样受体信号通路;hsa04622:RIG-I样受体信号通路;hsa04920:脂肪细胞因子信号通路;hsa03320:PPAR通路;hsa04152:AMPK通路。 图 4 基因簇KEGG的富集结果 |
本文结果与以往的研究相比,较为一致的是SPRR1B、CXCL10和IL-1B等基因被提示为枢纽基因[11-13]。这些基因的功能分别与角质形成细胞的分化、免疫细胞迁移和促炎活性相关,被认为是银屑病的重要节点基因[14-15]。此外,本研究有一些新的发现:①相比于单基因功能分析,簇集式的功能分析可以清晰、高效地发现和鉴定基因功能;②与传统治疗方法相比,单克隆抗体(特别是TNF-α、IL-17的抗体)治疗组的DEGs与银屑病组的DEGs匹配度更高,提示其在基因表达层面的作用更精确;③不同单克隆抗体治疗组基因表达的总体趋势基本一致,提示其存在协同途径的可能。
本文DEGs功能分析显示,基因簇1和2的功能分别与角化和有丝分裂周期有关。角质形成细胞增生过度以及分化障碍与银屑病有关,细胞周期相关产物在角质细胞的增殖中起重要作用[4, 14]。根据本文功能分析的结果以及基因相互作用的分析,簇1中的SPRR1B和S100A7以及簇2中的CCNB1和CDK1最有可能是银屑病皮损中细胞增殖的关键基因。趋化因子及相应的信号通路在银屑病的发病中起到一定作用,趋化因子如CXCL8被认为与中性粒细胞、单核细胞在表皮中聚集有关[7, 15],而包括CCL20和CXCL1等趋化因子在内的基因簇3被认为与趋化因子相关通路有密切的联系[16]。本文结果显示,趋化因子相关基因如CXCL9、CXCL10和CXCL8属于银屑病组的上调基因,基因簇4和5中的基因主要与IL-17通路相关。
本文研究所有治疗组中,依那西普治疗组的DEGs与银屑病组的匹配度最佳。TNF-α抑制剂是银屑病治疗中最基本的一类生物制剂。其治疗机制被认为是阻断白细胞,特别是辅助T细胞的活化,从而减轻表皮的炎症[17]。与以往研究较一致的是,依那西普调控的基因与角化过程、有丝分裂和趋化因子相关[18]。此外,依那西普能降低IL-17A相关基因的表达,而银屑病皮损组织中也发现了TNF参与IL-17A信号通路的依据[19]。本文研究中另一种TNF-α抗体英夫利昔单抗(infliximab)的治疗组由于样本数少未发现DEGs。
包括布罗达单抗、司库奇尤单抗和伊西贝单抗在内的IL-17抗体对皮损组织的基因表达有明显的作用。本文研究结果显示,银屑病组中几乎所有的DEGs均被布罗达单抗治疗组覆盖。银屑病皮损中,IL-17A已被证实可刺激角质形成细胞释放其他细胞因子,从而募集炎症细胞,并促进角质形成细胞的增殖[20-21]。从细胞因子信号转导的角度,司库奇尤单抗和伊西贝单抗为靶向IL-17A的抗体,而布罗达单抗阻断IL-17RA,IL-17RA是IL-17A和许多其他细胞因子的共同受体,故布罗达单抗治疗组涉及的DEGs更广泛[9, 20]。
本文基因交集分析显示,其他单克隆抗体中,乌司奴单抗(ustekinumab,IL-12/23抗体)组和古塞库单抗(IL-23抗体)组未发现与银屑病组有DEGs交集;糖皮质激素(glucocorticoids)、维A酸(treti-noin)、延胡索酸酯和甲氨蝶呤组中几乎没有可匹配的DEGs;UVB组的DEGs与银屑病组存在一个较小的交集,功能分析提示其与IL-17通路相关。
本文各治疗组的未匹配DEGs中也发现了一些与治疗相关的可能机制,司库奇尤单抗、伊西贝单抗和古塞库单抗在IL-17信号通路上有交集,其中编码炎症因子β-防御素的基因DEFB4A和DEFB4B在银屑病的发病中起到关键作用[22]。司库奇尤单抗、古塞库单抗和UVB治疗组中的DEGs在角化过程中存在共同作用,该组核心基因为KRT6C和SPRR2A,与表皮细胞的增生以及银屑病发病均有关[23]。此外,维A酸作为银屑病的治疗药物已有多年历史,GO和KEGG分析的结果提示,依那西普和古塞库单抗治疗组的DEGs在维A酸代谢方面有相同的作用,故维A酸与单克隆抗体也可能存在相互作用[24]。
综上所述,单克隆抗体治疗银屑病显示出较传统疗法更优的疗效。特别是TNF-α和IL-17抗体治疗后组织的基因表达情况与银屑病组有较高的一致性和符合率。本研究初步揭示了银屑病发病及治疗中的部分关键基因和基因簇。不同单克隆抗体之间以及与其他疗法之间的协同作用仍需在未来的研究中进一步探索。
| [1] |
ARMSTRONG A W, READ C. Pathophysiology, clinical presentation, and treatment of psoriasis: a review[J]. JAMA, 2020, 323(19): 1945-1960. DOI:10.1001/jama.2020.4006 |
| [2] |
MICHALEK I M, LORING B, JOHN S M. A systematic review of worldwide epidemiology of psoriasis[J]. Journal of the European Academy of Dermatology and Venereology: JEADV, 2017, 31(2): 205-212. DOI:10.1111/jdv.13854 |
| [3] |
BAKSHI H, NAGPAL M, SINGH M, et al. Treatment of psoriasis: a comprehensive review of entire therapies[J]. Current Drug Safety, 2020, 15(2): 82-104. |
| [4] |
LOWES M A, BOWCOCK A M, KRUEGER J G. Pathogenesis and therapy of psoriasis[J]. Nature, 2007, 445(7130): 866-873. DOI:10.1038/nature05663 |
| [5] |
LI A H, LI W W, YU X Q, et al. Bioinformatic analysis and translational validation of psoriasis candidate genes for precision medicine[J]. Clinical, Cosmetic and Investigational Dermatology, 2022, 15: 1447-1458. DOI:10.2147/CCID.S378143 |
| [6] |
WANG H L, CHEN W J, HE J, et al. Network analysis of potential risk genes for psoriasis[J]. Hereditas, 2021, 158(1): 21. DOI:10.1186/s41065-021-00186-w |
| [7] |
YANG Y, XIE S Q, JIANG W C, et al. Discovering novel biomarkers associated with the pathogenesis of psoriasis: evidence from bioinformatic analysis[J]. International Journal of General Medicine, 2022, 15: 2817-2833. DOI:10.2147/IJGM.S354985 |
| [8] |
RUSSELL C B, RAND H, BIGLER J, et al. Gene expression profiles normalized in psoriatic skin by treatment with brodalumab, a human anti-IL-17 receptor monoclonal antibody[J]. Journal of Immunology, 2014, 192(8): 3828-3836. DOI:10.4049/jimmunol.1301737 |
| [9] |
TOMALIN L E, RUSSELL C B, GARCET S, et al. Short-term transcriptional response to IL-17 receptor-a antagonism in the treatment of psoriasis[J]. The Journal of Allergy and Clinical Immunology, 2020, 145(3): 922-932. DOI:10.1016/j.jaci.2019.10.041 |
| [10] |
ZABA L C, SUÁREZ-FARIÑAS M, FUENTES-DUCULAN J, et al. Effective treatment of psoriasis with etanercept is linked to suppression of IL-17 signaling, not immediate response TNF genes[J]. Journal of Allergy and Clinical Immunology, 2009, 124(5): 1022-1030.e395. DOI:10.1016/j.jaci.2009.08.046 |
| [11] |
AYDIN B, ARGA K Y, KARADAG A S. Omics-driven biomarkers of psoriasis: recent insights, current challenges, and future prospects[J]. Clinical, Cosmetic and Investigational Dermatology, 2020, 13: 611-625. DOI:10.2147/CCID.S227896 |
| [12] |
FERRARI S M, RUFFILLI I, COLACI M, et al. CXCL10 in psoriasis[J]. Advances in Medical Sciences, 2015, 60(2): 349-354. DOI:10.1016/j.advms.2015.07.011 |
| [13] |
BUERGER C, RICHTER B, WOTH K, et al. Interleukin-1β interferes with epidermal homeostasis through induction of insulin resistance: implications for psoriasis pathogenesis[J]. The Journal of Investigative Dermatology, 2012, 132(9): 2206-2214. DOI:10.1038/jid.2012.123 |
| [14] |
LUO Y Q, LUO Y Y, CHANG J, et al. Identification of candidate biomarkers and pathways associated with psoriasis using bioinformatics analysis[J]. Hereditas, 2020, 157(1): 30. DOI:10.1186/s41065-020-00141-1 |
| [15] |
NICKOLOFF B J, XIN H, NESTLE F O, et al. The cytokine and chemokine network in psoriasis[J]. Clinics in Dermatology, 2007, 25(6): 568-573. DOI:10.1016/j.clindermatol.2007.08.011 |
| [16] |
SU W X, WEI Y Q, HUANG B, et al. Identification of hub genes and immune infiltration in psoriasis by bioinformatics method[J]. Frontiers in Genetics, 2021, 12: 606065. DOI:10.3389/fgene.2021.606065 |
| [17] |
GOTTLIEB A B, CHAMIAN F, MASUD S, et al. TNF inhibition rapidly down-regulates multiple proinflammatory pathways in psoriasis plaques[J]. Journal of Immunology, 2005, 175(4): 2721-2729. DOI:10.4049/jimmunol.175.4.2721 |
| [18] |
ANTONATOS C, PANOUTSOPOULOU M, GEORGAKILAS G K, et al. Gene expression meta-analysis of potential shared and unique pathways between autoimmune diseases under anti-TNFα therapy[J]. Genes, 2022, 13(5): 776. DOI:10.3390/genes13050776 |
| [19] |
JOHNSTON A, GUZMAN A M, SWINDELL W R, et al. Early tissue responses in psoriasis to the antitumour necrosis factor-α biologic etanercept suggest reduced interleukin-17 receptor expression and signalling[J]. The British Journal of Dermatology, 2014, 171(1): 97-107. DOI:10.1111/bjd.12937 |
| [20] |
TOLLENAERE M A X, HEBSGAARD J, EWALD D A, et al. Signalling of multiple interleukin (IL)-17 family cytokines via IL-17 receptor A drives psoriasis-related inflammatory pathways[J]. The British Journal of Dermatology, 2021, 185(3): 585-594. DOI:10.1111/bjd.20090 |
| [21] |
JOHNSTON A, FRITZ Y, DAWES S M, et al. Keratinocyte overexpression of IL-17C promotes psoriasiform skin inflammation[J]. Journal of Immunology, 2013, 190(5): 2252-2262. DOI:10.4049/jimmunol.1201505 |
| [22] |
JOHANSEN C, BERTELSEN T, LJUNGBERG C, et al. Characterization of TNF-α- and IL-17A-mediated synergistic induction of DEFB4 gene expression in human keratinocytes through IκBζ[J]. The Journal of Investigative Dermatology, 2016, 136(8): 1608-1616. DOI:10.1016/j.jid.2016.04.012 |
| [23] |
GAO L H, DOU J H, ZHANG B, et al. Ozone therapy promotes the differentiation of basal keratinocytes via increasing Tp63-mediated transcription of KRT10 to improve psoriasis[J]. Journal of Cellular and Molecular Medicine, 2020, 24(8): 4819-4829. DOI:10.1111/jcmm.15160 |
| [24] |
CHEN J, MA Y Q, TAO Y Y, et al. Formulation and evaluation of a topical liposomal gel containing a combination of ze-doary turmeric oil and tretinoin for psoriasis activity[J]. Journal of Liposome Research, 2021, 31(2): 130-144. DOI:10.1080/08982104.2020.1748646 |
 2025, Vol. 61
2025, Vol. 61

