染色体的准确分离能够确保遗传物质平均分配到两个子细胞中[1]。人源Aurora B基因定位于染色体17号染色体短臂13区[2],其表达产物参与染色体乘客复合体(CPC)的形成和染色体的调控[3]。Aurora B激酶能够对染色体上多种蛋白质进行磷酸化[4-5]并发生自磷酸化[6],而PP2A-B56和PP1等蛋白磷酸酶可以通过对Aurora B激酶底物去磷酸化调控有丝分裂的正常进行[7-9]。Hesperadin是一种Aurora B激酶抑制剂,可抑制其底物组蛋白H3 S10发生磷酸化[10],而冈田软海绵酸(OA)能够抑制磷酸酶活性。最近的研究表明,染色体表面的多种蛋白质被有丝分裂激酶磷酸化从而影响染色体的形态变化[11]。本实验通过免疫荧光技术、活细胞成像等多种实验技术,探究Aurora B激酶对有丝分裂染色体形态变化的影响及其作用机制。
1 材料与方法 1.1 实验材料HeLa细胞购自中国科学院上海生物研究所细胞资源中心。持续表达H2B-CFP的HeLa细胞系由课题组建立。DMEM培养液、L-15培养液(美国Gibco公司),胎牛血清(美国HyClone公司),Thymidine(美国Sigma公司),Hesperadin、DMA、OA(美国Selleck公司),鼠抗人Tubulin单克隆抗体、兔抗人H3 S10多克隆抗体(英国Abcam公司),鼠抗人Cyclin B1单克隆抗体(美国Santa Cruz Biotechnology公司),鼠抗人GAPDH单克隆抗体(美国Proteintech公司),DAPI、荧光二抗Alexa Fluro Dyes 488(美国Thermo公司)。
1.2 实验方法 1.2.1 细胞培养将HeLa细胞置于添加体积分数0.10胎牛血清和10 g/L青链霉素溶液的DMEM培养液中,在37 ℃、含体积分数0.05 CO2的细胞培养箱中培养。
1.2.2 Western blot检测组蛋白H3 S10的磷酸化水平取对数生长期的HeLa细胞,以每孔约5×105个细胞的密度铺在6孔板中,同时加入40 μL浓度为0.6 g/L的Thymidine进行细胞同步化处理。18 h后将Thymidine用PBS洗掉,加入1 μL浓度为1 μg/L的DMA完全培养液过夜。将细胞分为A、B组,分别加入2 μL的二甲基亚砜(DMSO)和Hesperadin(浓度50 μg/L)处理1 h,收集细胞并用RIPA裂解液提取两组细胞总蛋白。应用BCA试剂盒检测蛋白质浓度,加入10×蛋白上样缓冲液100 ℃加热10 min使蛋白变性。配制SDS-PAGE凝胶进行电泳、转膜,加入50 g/L的脱脂奶粉室温封闭1 h,一抗Cyclin B1(1∶200)、H3 S10(1∶1 000)和GAPDH(1∶1 000)4 ℃摇床孵育过夜。然后使用TBST洗膜3次,加入相应种属二抗(1∶5 000)室温孵育1 h,重复上述洗膜步骤3次;配制ECL化学发光液显影,检测两组细胞Aurora B激酶底物H3 S10的磷酸化水平。以GAPDH条带结果为内参照,使用Image J软件分析蛋白条带灰度值,以H3 S10灰度值/GAPDH灰度值计算两组细胞H3 S10蛋白相对表达量。实验重复3次,结果取均值。
1.2.3 免疫荧光实验检测染色体的形态变化6孔板中每孔分别放入1个爬片,按照1.2.2的方法处理和分组,每组设3个孔。分别在药物处理30、60、90 min时弃掉培养液,PBS洗2次。每孔分别加入2 mL的40 g/L多聚甲醛,室温固定细胞20 min。使用体积分数0.005 Triton X-100/PBS给细胞膜打孔10 min,以PBST洗涤细胞2次,每次5 min。用镊子将爬片取出放到湿盒中, 加入体积分数0.03 BSA/PBST室温封闭1 h。吸掉封闭液,在爬片上滴加一抗Tubulin(1∶400),室温孵育3 h或者4 ℃过夜。将爬片置于PBST中洗3次,每次5 min。使用DAPI(1∶1 000)和荧光二抗Alexa Fluro Dyes 488(1∶1 000)室温染色1 h,重复上述洗片步骤。待爬片干燥后,滴加5 μL封片剂封片。使用徕卡Thunder高分辨率显微成像系统观察图像,计数两组细胞中染色体环状结构的细胞数量。
1.2.4 高分辨率活细胞成像实验观察染色体动态变化将稳定表达H2B-CFP的HeLa细胞系细胞接种在6孔板并进行同步化处理,24 h后消化细胞并按照7∶1的比例铺在含有新鲜培养液的活细胞培养皿中,12 h后加入DMA处理12 h。检测前将两组细胞培养液换为分别添加DMSO或Hesperadin的L-15培养液,将加入药物前记为“0”时刻。使用40倍物镜连续采集图像30 min,间隔5 min记录一次CFP信号,分析并提取所需要的静态图像。
1.2.5 细胞染色体分离实验检测细胞染色体聚集将两组细胞培养液中分别加入DMSO或者Hesperadin处理1 h,将收集的有丝分裂细胞用PBS洗1遍并进行细胞计数。将细胞稀释到109/L,加入低渗缓冲液37 ℃处理10 min。在涂片机中放入载玻片和200 μL细胞悬液,1 300 r/min离心10 min。滴加40 g/L多聚甲醛固定细胞15 min,随后加入体积分数0.005 Triton X-100/PBS细胞打孔5 min。DAPI染色,显微成像系统中使用100倍物镜拍摄图片,观察抑制Aurora B激酶对染色体形态的影响。
1.2.6 免疫荧光实验检测蛋白磷酸酶对染色体形态变化的影响将同步化处理后的HeLa细胞分为A组、B组以及C组并加入DMA过夜处理。A组和B组细胞按1.2.2方法进行处理,C组细胞同时加入Hesperadin和OA,90 min后固定细胞进行免疫荧光染色,观察抑制磷酸酶对染色体聚集细胞的影响。
1.2.7 siRNA筛选Aurora B激酶的底物蛋白根据相关研究结果选择30个Aurora B激酶可能的相互作用蛋白[10](大部分已被证明与染色体相关),由上海吉玛基因公司合成针对不同蛋白的siRNA oligo。在HeLa细胞中分别转染Luciferase和不同的siRNA,加入Hesperadin处理1 h,固定细胞并使用DAPI和Tubulin抗体染色,荧光显微镜下计数染色体环状结构细胞数量。
1.3 统计学方法使用SPSS 22.0、Graphpad Prism 8.0软件进行统计学分析。计量资料以x±s表示,两组数据比较采用t检验;多组数据比较采用析因设计方差分析及单因素方差分析(ANOVA),组间两两比较采用LSD-t法。以P < 0.05为差异有统计学意义。
2 结果 2.1 抑制Aurora B激酶活性对组蛋白H3 S10磷酸化水平影响Western blot检测显示,A组、B组H3 S10蛋白磷酸化水平分别为1.14±0.06、0.22±0.04,两组比较差异有显著性(t=23.58,P < 0.05)。见图 1。
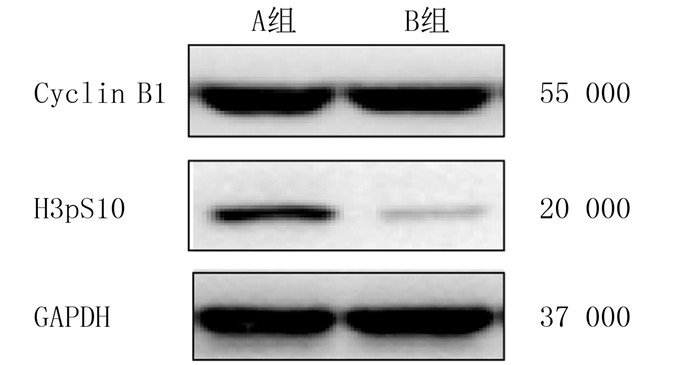 |
| 图 1 Western blot方法检测两组H3 S10的磷酸化水平 |
免疫荧光染色显示,A组细胞染色体排列分散,在未分离的中心体周围呈玫瑰花环状圆形结构;B组有丝分裂细胞染色体聚集,堆叠在一起形成特异性环状结构(图 2A)。析因设计方差分析显示,时间、组别及二者交互作用对染色体聚集细胞数量影响差异均有统计学意义(F组别=1 657.56,F时间=17.48,F时间×组别=14.94,P < 0.05)。单独效应结果显示,A组不同时间点染色体聚集细胞数量差异无统计学意义(F=0.053,P>0.05),B组不同时间点染色体聚集细胞数量差异有统计学意义(F=32.36,P < 0.05)。与A组相比,B组在30、60和90 min时染色体聚集细胞数量显著增多,差异有统计学意义(F=379.28~738.73,P < 0.05)。组内比较结果显示,B组在60、90 min染色体聚集细胞数量较30 min明显增多(P < 0.05)(图 2B)。
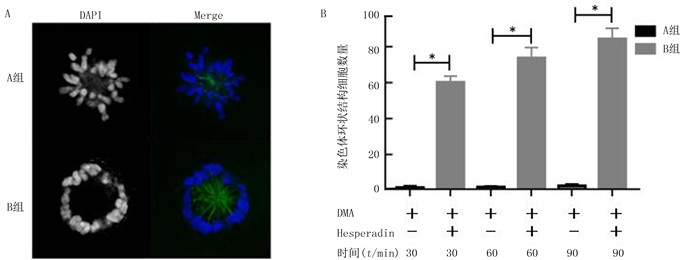 |
| A:两组细胞染色体的静态图片。DAPI染色呈蓝色,Tubulin染色呈绿色,100倍。B:两组细胞不同时间染色体环状结构细胞数量比较。与A组相比,*P < 0.05。 图 2 免疫荧光实验检测两组细胞染色体形态变化 |
A组细胞有丝分裂过程中染色体排列分散,围绕中心体呈玫瑰花环状圆形结构;B组细胞加入Hesperadin后,染色体逐渐聚集,呈特异性环状结构(图 3)。
 |
| 放大40倍。 图 3 活细胞成像实验观察两组细胞染色体动态变化 |
荧光显微镜下观察显示,A组细胞整体与放大图片染色体排列较为分散;B组细胞染色体整体较为聚集,局部放大观察发现,染色体之间出现黏连现象(图 4)。
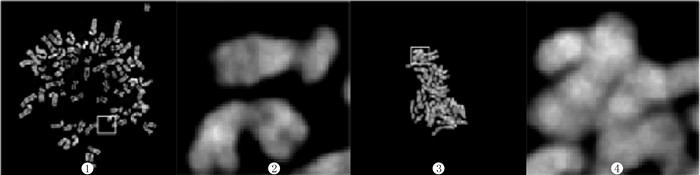 |
| ①、②为A组,③、④为B组,①、③放大100倍,②、④放大200倍。 图 4 两组细胞染色体分离观察 |
免疫荧光检测显示,A组、B组和C组染色体聚集细胞数量分别为(1.33±0.57)、(82.67±2.52)、(32.00±3.00)个,各组间比较差异有显著性(n=100,F=969.2,P < 0.05)。两两比较显示,C组染色体聚集的细胞数量明显低于B组(P < 0.05),高于A组(P < 0.05),说明抑制Aurora B激酶会导致磷酸酶发挥去磷酸化作用,促进染色体的聚集。
2.4 Aurora B激酶底物蛋白筛选免疫荧光结果显示,MCM7组与Luciferase组染色体聚集细胞数量分别为77.33 ± 1.20、16.33 ± 0.88,两组比较差异有显著统计学意义(t=40.92,P < 0.05),表明MCM7蛋白被RNA干扰后,抑制Aurora B激酶导致染色体聚集细胞数量明显减少(图 5)。
 |
| 图 5 siRNA筛选Aurora B激酶影响染色体形态变化的底物蛋白 |
细胞有丝分裂的正常进行是保证染色体准确分离的前提[12]。一系列的丝氨酸/苏氨酸激酶在有丝分裂过程中发挥重要作用,其异常表达可能会导致乳癌以及其他癌症的发生和发展[13-14]。近年来的研究表明,Aurora B激酶过度表达与肿瘤的发生发展有关,并被认为是一种潜在的生物标志物[15]。由于Aurora B激酶调控有丝分裂的分子机制目前尚不完全清楚,因此,探讨Aurora B激酶影响染色体分离的分子机制对于肿瘤的诊断、靶向治疗具有重要意义。
本研究首次探究了Aurora B激酶对有丝分裂染色体形态变化的影响。Western blot实验结果显示,在细胞有丝分裂期,抑制Aurora B激酶活性可引起组蛋白H3 S10磷酸化水平降低;免疫荧光实验和活细胞成像以及染色体分离实验结果表明,抑制Aurora B激酶能够促进染色体的聚集。为进一步探究Aurora B激酶调控染色体形态变化的分子机制,本研究在抑制Aurora B激酶的基础上同时加入磷酸酶抑制剂观察染色体形态变化,结果显示抑制Aurora B激酶会导致磷酸酶发挥去磷酸化作用,促进染色体的聚集;进一步通过siRNA筛选发现,染色体上可能存在MCM7等多种Aurora B激酶的底物蛋白影响染色体的形态变化。
综上所述,有丝分裂激酶Aurora B与蛋白磷酸酶协同调控有丝分裂染色体的形态变化。本课题下一步将通过免疫共沉淀等多种实验方法,鉴定本文筛选出的蛋白是否为有丝分裂激酶Aurora B的底物,并进一步探究其作用位点和具体的分子机制,以了解染色体发生形态变化的生理意义,为肿瘤的临床诊断和靶点治疗提供理论基础。
| [1] |
KITAGAWA M, LEE S H. The chromosomal passenger complex (CPC) as a key orchestrator of orderly mitotic exit and cytokinesis[J]. Frontiers in Cell and Developmental Biology, 2015, 3: 14. |
| [2] |
KOMAKI S, TAKEUCHI H, HAMAMURA Y, et al. Functional analysis of the plant chromosomal passenger complex[J]. Plant Physiology, 2020, 183(4): 1586-1599. DOI:10.1104/pp.20.00344 |
| [3] |
SALIMIAN K J, BALLISTER E R, SMOAK E M, et al. Feedback control in sensing chromosome biorientation by the Aurora B kinase[J]. Current Biology: CB, 2011, 21(13): 1158-1165. DOI:10.1016/j.cub.2011.06.015 |
| [4] |
SHIMADA M, GOSHIMA T, MATSUO H, et al. Essential role of autoactivation circuitry on aurora B-mediated H2AX-pS121 in mitosis[J]. Nature Communications, 2016, 7: 12059. DOI:10.1038/ncomms12059 |
| [5] |
MANZIONE M G, ROMBOUTS J, STEKLOV M, et al. Co-regulation of the antagonistic RepoMan: Aurora-B pair in proliferating cells[J]. Molecular Biology of the Cell, 2020, 31(6): 419-438. DOI:10.1091/mbc.E19-12-0698 |
| [6] |
YASUI Y, URANO T, KAWAJIRI A, et al. Autophosphorylation of a newly identified site of Aurora-B is indispensable for cytokinesis[J]. The Journal of Biological Chemistry, 2004, 279(13): 12997-13003. DOI:10.1074/jbc.M311128200 |
| [7] |
LIU D, VLEUGEL M, BACKER C B, et al. Regulated targeting of protein phosphatase 1 to the outer kinetochore by KNL1 opposes Aurora B kinase[J]. The Journal of Cell Biology, 2010, 188(6): 809-820. DOI:10.1083/jcb.201001006 |
| [8] |
NASA I, RUSIN S F, KETTENBACH A N, et al. Aurora B opposes PP1 function in mitosis by phosphorylating the conserved PP1-binding RVxF motif in PP1 regulatory proteins[J]. Science Signaling, 2018, 11(530): eaai8669. DOI:10.1126/scisignal.aai8669 |
| [9] |
HAUF S, COLE R W, LATERRA S, et al. The small molecule Hesperadin reveals a role for Aurora B in correcting kinetochore-microtubule attachment and in maintaining the spindle assembly checkpoint[J]. The Journal of Cell Biology, 2003, 161(2): 281-294. DOI:10.1083/jcb.200208092 |
| [10] |
HENGEVELD R C, HERTZ N T, VROMANS M J, et al. Development of a chemical genetic approach for human aurora B kinase identifies novel substrates of the chromosomal passenger complex[J]. Molecular & Cellular Proteomics: MCP, 2012, 11(5): 47-59. |
| [11] |
GARTNER M, SUNDER-PLASSMANN N, SEILER J, et al. Development and biological evaluation of potent and specific inhibitors of mitotic Kinesin Eg5[J]. ChemBioChem: a European Journal of Chemical Biology, 2005, 6(7): 1173-1177. DOI:10.1002/cbic.200500005 |
| [12] |
SHAKEEL I, BASHEER N, HASAN G M, et al. Polo-like Kinase 1 as an emerging drug target: structure, function and therapeutic implications[J]. Journal of Drug Targeting, 2021, 29(2): 168-184. DOI:10.1080/1061186X.2020.1818760 |
| [13] |
HONMA K, NAKANISHI R, NAKANOKO T, et al. Contribution of Aurora-A and -B expression to DNA aneuploidy in gastric cancers[J]. Surgery Today, 2014, 44(3): 454-461. DOI:10.1007/s00595-013-0581-x |
| [14] |
WU X, LIU J M, SONG H H, et al. Aurora-B knockdown inhibits osteosarcoma metastasis by inducing autophagy via the mTOR/ULK1 pathway[J]. Cancer Cell International, 2020, 20(1): 575. |
| [15] |
GALETTA D, CORTES-DERICKS L. Promising therapy in lung cancer: spotlight on aurora kinases[J]. Cancers, 2020, 12(11): E3371. DOI:10.3390/cancers12113371 |
 2023, Vol. 59
2023, Vol. 59

