2. 青岛大学附属医院耳鼻咽喉头颈外科, 山东 青岛 266071
鼻咽癌属于高度恶性的头颈部肿瘤,早期诊断困难且缺乏特异性的检测指标[1]。放射治疗是非转移鼻咽癌的主要治疗手段[2],但放射抵抗的出现会引起局部复发以及远处转移,导致治疗失败[3-4]。因此寻找新的治疗靶点以改善病人的预后具有重要的意义。microRNAs(miRNAs)可以通过互补序列与靶标mRNA分子结合从而抑制mRNA的翻译[5]。miRNAs作为遗传调节因子,在鼻咽癌的发生发展中至关重要[6-10]。研究表明,miR-1185-5p在鼻咽癌中存在差异表达,但其作用尚未阐明[11]。磷脂酰肌醇转运蛋白β (PITPNB) 3′-非编码区(UTR)内存在与miR-1185-5p碱基互补配对的序列,推测miR-1185-5p可能对PITPNB具有调控作用。本研究探讨miR-1185-5p对鼻咽癌细胞增殖和迁移的调控作用,并以PITPNB为靶点探讨其下游机制,旨在为鼻咽癌的治疗提供新方向。
1 材料与方法 1.1 TCGA数据库检索及PITPNB与miR-1185-5p结合位点的预测检索癌症基因组图谱(TCGA)数据库中43组配对的头颈部鳞状细胞癌样本及其癌旁正常样本的转录组数据与临床数据,应用“ggplot2”R包分析其中miR-1185-5p和PITPNB的差异表达,应用“survminer”R包分析PITPNB表达与病人总生存率间的关系;应用TargetScan(http://www.targetscan.org/)预测miR-1185-5p与PITPNB之间的结合位点。
1.2 细胞培养应用含体积分数0.10胎牛血清(以色列BI公司)的RPMI-1640培养液(大连美仑公司),37 ℃条件下,在含体积分数0.05的CO2培养箱中培养鼻咽癌CNE-2细胞(上海通派公司)。
1.3 质粒构建及转染应用T4 DNA Ligase(南京诺唯赞公司生产)将人PITPNB(NM_001284277)的cDNA克隆至pPB-CAG-pgk-hph(湖南丰晖公司)转座子载体,构建PITPNB过表达质粒(OE-PITPNB),以转座子载体作为空载对照质粒(Control)。PITPNB的3′UTR被合成并插入pGL6荧光素酶报告载体(北京碧云天公司),用以构建PITPNB野生型共表达载体(PITPNB-WT)。将PITPNB的3′UTR(UAUCCUC)与miR-1185-5p的结合位点突变为AUAGGAG,并插入pGL6荧光素酶报告载体,用以构建PITPNB突变型共表达载体(PITPNB-MUT)。将CNE-2细胞接种至相应孔板,当细胞融合至70%~90%时转染。miRNAs与质粒的转染时间及用量参考PEI(美国Polysciences公司)转染试剂说明书。
1.4 RT-PCR方法检测miR-1185-5p和PITPNB mRNA的表达应用RNA isolater(南京诺唯赞公司)提取细胞总RNA,应用EvoM-MLV反转录试剂盒(湖南艾科瑞公司)将RNA逆转录为cDNA,SYBR Green Pro Taq HS(湖南艾科瑞公司)试剂盒进行RT-PCR扩增,根据试剂盒说明书设置反应的体系及条件。将CNE-2细胞分为4组,分别转染mimic NC、miR-1185-5p mimic、inhibitor NC和miR-1185-5p inhibitor,48 h后应用RT-PCR方法分别检测各组miR-1185-5p以及PITPNB mRNA的表达水平。再将CNE-2细胞分为2组,分别转染空载对照质粒(Control)和OE-PITPNB,24 h后应用RT-PCR方法检测各组PITPNB mRNA表达。以U6为内参检测miR-1185-5p的相对表达量,以GAPDH为内参检测PITPNB mRNA的相对表达量。RT-PCR引物及其序列见表 1。
| 表 1 RT-PCR引物及其序列 |
|
|
将CNE-2细胞以每孔4 000个的密度接种于96孔板,细胞分组及处理同1.4,按照试剂盒说明书要求加入CCK-8试剂(上海翌圣公司),37 ℃孵育30 min后应用酶标仪检测450 nm波长下的吸光度值。每组均设置4个复孔。
1.6 划痕实验将CNE-2细胞接种至6孔板,当细胞融合至70%时,按1.4方法进行分组及转染,48 h后使用200 μL无菌枪头划痕,PBS润洗细胞后加入无血清RPMI-1640培养液再置于培养箱中,从0 h开始每隔12 h在倒置显微镜下采集图像,应用Image J软件分析图像。
1.7 双荧光素酶报告基因检测将CNE-2细胞接种于12孔板中的4个孔,分别转染mimic NC+PITPNB-WT+海肾荧光报告载体(北京碧云天公司生产)、miR-1185-5p mimic+PITPNB-WT+海肾荧光报告载体、mimic NC+PITPNB-MUT+海肾荧光报告载体、miR-1185-5p mimic+PITPNB-MUT+海肾荧光报告载体。转染48 h后裂解细胞收集上清,应用Dual-Luciferase(Promega)双荧光素酶报告基因系统检测荧光素酶活性。
1.8 统计学方法应用GraphPad 8.3软件进行统计分析。正态分布计量资料以x±s形式表示,两组间比较应用t检验,多组间比较应用双因素方差分析;非正态分布计量资料以M(Q1, Q3)形式表示,数据间比较采用Wilcoxon符号秩和检验;生存分析应用Log-rank检验。以P < 0.05为差异有统计学意义。
2 结果 2.1 miR-1185-5p对鼻咽癌细胞增殖和迁移的影响对TCGA数据库中的43对头颈鳞癌和癌旁正常组织样本分析显示,头颈鳞癌组织中miR-1185-5p的表达显著低于癌旁正常组织(t=-2.17, P < 0.05)。见图 1A。RT-PCR检测显示,与mimic NC组细胞相比,转染miR-1185-5p mimic可显著上调miR-1185-5p的表达(t=96.18,P < 0.001);与inhibitor NC组相比,转染miR-1185-5p inhibitor抑制了miR-1185-5p的表达(t=4.98,P < 0.05)。见图 1B。CCK-8实验结果显示,mimic NC组和miR-1185-5p mimic组相比较,CNE-2细胞增殖能力存在时间和组别的交互作用(F=6.079,P < 0.05),随着培养时间的延长(F=246.800,P < 0.001),miR-1185-5p mimic组CNE-2细胞的增殖能力较mimic NC组降低(F=8.086,P < 0.05)。同样,inhibitor NC组和miR-1185-5p inhibitor组亦存在时间和组别交互作用(F=12.360,P < 0.01),随着培养时间延长(F=121.600,P < 0.001),miR-1185-5p inhibitor组细胞增殖能力较inhibitor NC组细胞增高(F=11.160,P < 0.01)。见图 1C。划痕实验结果显示,miR-1185-5p mimic组CNE-2细胞的迁移能力较mimic NC组降低(t=12.83,P < 0.05),而miR-1185-5p inhibitor组CNE-2细胞的迁移能力较inhibitor NC组升高(t=12.78, P < 0.05)。见图 1D、E。
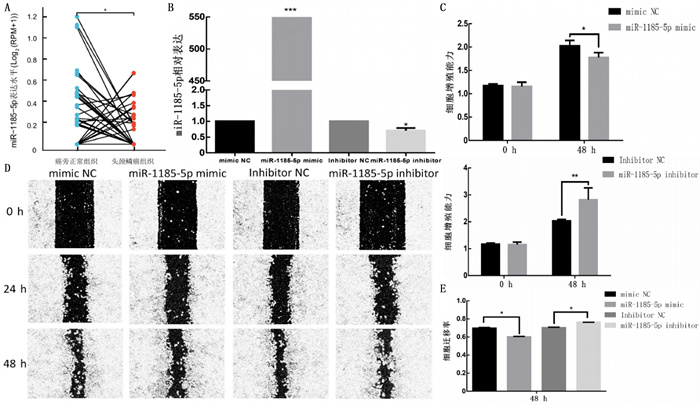 |
| A:TCGA数据库中miR-1185-5p的相对表达;B:转染mimic NC、miR-1185-5p mimic、inhibitor NC和miR-1185-5p inhibitor后miR-1185-5p的相对表达;C:miR-1185-5p对CNE-2细胞增殖的影响;D、E:miR-1185-5p对CNE-2细胞迁移的影响。*P < 0.05, **P < 0.01, ***P < 0.001。 图 1 miR-1185-5p对鼻咽癌CNE-2细胞增殖和迁移的调控作用 |
TargetScan数据库分析显示,miR-1185-5p与PITPNB的3′UTR之间存在结合位点(图 2A)。应用TCGA数据库分析表明,PITPNB在头颈鳞癌组织中高表达(Z=70,P < 0.001)。见图 2B。生存分析显示,PITPNB高表达的头颈部鳞状细胞癌病人的总生存率较低(χ2=6.05,P < 0.05)。见图 2C。
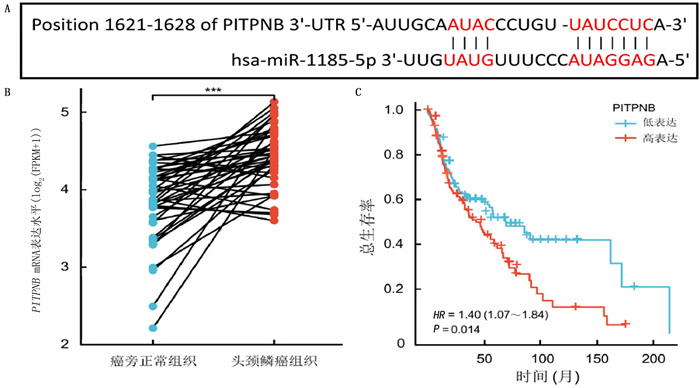 |
| A:PITPNB的3′UTR内miR-1185-5p结合位点示意图;B:TCGA数据库中PITPNB mRNA的相对表达;C:不同PITPNB表达水平的头颈部鳞状细胞癌病人的生存分析。***P < 0.001。 图 2 miR-1185-5p与PITPNB靶向结合关系的预测 |
与Control组相比,转染OE-PITPNB后鼻咽癌CNE-2细胞PITPNB的表达升高(t=6.55,P < 0.01)。见图 3A。CCK-8实验显示,CNE-2细胞的增殖能力存在时间和组别的交互作用(F=12.000,P < 0.01),当培养时间至24 h时(F=126.300,P < 0.001),OE-PITPNB组CNE-2细胞的增殖能力较Control组显著提高(F=18.010,P < 0.01)。见图 3B。划痕实验显示,当培养时间至48 h时,Control组细胞迁移率为(51.86±0.82)%,OE-PITPNB组细胞迁移率为(70.41±0.56)%,OE-PITPNB组CNE-2细胞的迁移能力较Control组增强(t=13.39, P < 0.05)。见图 3C。
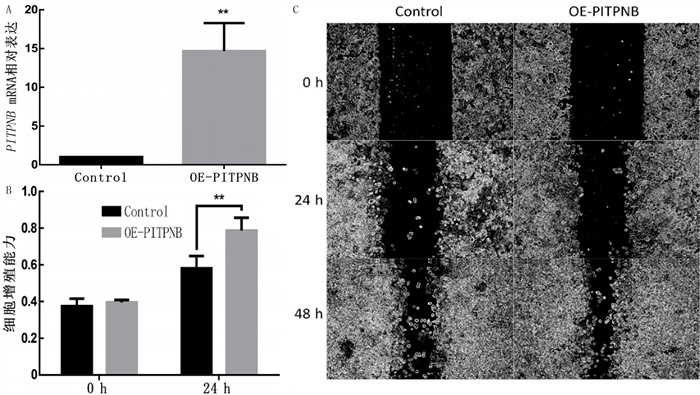 |
| A:两组CNE-2细胞中PITPNB mRNA的相对表达;B:PITPNB对CNE-2细胞增殖的影响;C:PITPNB对CNE-2细胞迁移的影响。*P < 0.05, **P < 0.01, ***P < 0.001。 图 3 PITPNB对鼻咽癌CNE-2细胞增殖和迁移的调控作用 |
RT-PCR结果显示,与mimic NC组相比,miR-1185-5p mimic组PITPNB mRNA的表达显著降低(t=53.40,P < 0.001),而miR-1185-5p inhibitor组PITPNB mRNA的表达较inhibitor NC组升高(t=5.49,P < 0.05)。双荧光素酶报告基因检测结果显示,与mimic NC组相比,miR-1185-5p mimic组PITPNB-WT荧光素酶的活性降低(t=8.05,P < 0.01),而PITPNB-MUT荧光素酶活性差异无显著性(t=1.65,P>0.05)。见图 4。
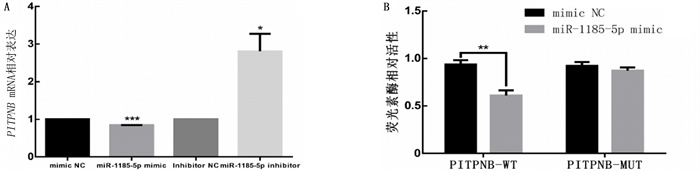 |
| A:各组CNE-2细胞中PITPNB mRNA的相对表达;B:相对荧光素酶活性。*P < 0.05, **P < 0.01, ***P < 0.001。 图 4 miR-1185-5p靶向PITPNB对其表达影响 |
鼻咽癌是一种与EB病毒感染有关的恶性肿瘤,占头颈部鳞癌的70%以上[12-13]。由于鼻咽癌早期缺乏明显的临床体征,超过70%的病人初诊即已经属于晚期。同步放化疗是晚期鼻咽癌的标准治疗方法[14]。虽然放疗和综合化疗策略的进步使原发性鼻咽癌病人的预后有了很大的改善,但鼻咽癌的复发率仍高达82%[15-17],复发后的再治疗是临床上存在的难题。EB病毒抗体血清学检测是目前临床上广泛应用的鼻咽癌筛查方法,但由于存在较高的假阳性率,阳性人群需反复随访,造成检查成本增高,给病人带来严重的心理负担。因此,寻找新的生物标志物和治疗靶点至关重要。
miRNAs是一种小型非编码RNA,通过识别同源序列和干扰转录、翻译或表观遗传过程来调节基因表达[5]。miRNAs可参与多种细胞的生物学过程并发挥作用,包括细胞周期调节、应激反应、炎症、分化、增殖、侵袭和凋亡等[18-22]。研究表明,miRNAs功能失调可导致包括鼻咽癌在内的多种肿瘤的发生[23-24]。因此,识别miRNAs、靶标及其调控网络对于阐明miRNAs的正常生物学过程及其在肿瘤发生发展中的作用至关重要[25]。相关研究表明,miR-1185在不同肿瘤中均低表达,其中miR-1185在结直肠癌细胞中表达下调,并在mTOR通路中发挥作用[26];在人类乳癌中miR-1185也呈低表达,且与病人预后负相关[27]。本研究通过对TCGA数据库中43对头颈部鳞状细胞癌和癌旁正常组织的转录组数据分析发现,与癌旁正常组织相比较,头颈部鳞状细胞癌组织miR-1185-5p低表达,提示miR-1185-5p可能作为肿瘤抑制因子在头颈部鳞状细胞癌中发挥作用。本研究通过转染miR-1185-5p mimic和miR-1185-5p inhibitor分别升高和抑制miR-1185-5p在鼻咽癌CNE-2细胞中的表达,通过CCK-8实验和划痕实验探讨miR-1185-5p在CNE-2细胞中的作用机制。结果显示,上调miR-1185-5p表达抑制了CNE-2细胞的增殖和迁移,而抑制miR-1185-5p表达对CNE-2细胞的增殖和迁移具有促进作用。提示miR-1185-5p作为抑癌因子参与鼻咽癌的生物学过程。
PITPNB可以催化磷脂酰肌醇和磷脂酰胆碱在细胞膜间的转移,这种转运活性是包被蛋白复合体Ⅰ介导的从高尔基体到内质网的逆行转运所必需的。目前,PITPNB在肿瘤中作用的研究还未广泛开展。本文生物信息学分析显示,PITPNB在头颈部鳞状细胞癌中高表达,且高表达PITPNB的病人预后较差;进一步探究PITPNB对CNE-2细胞生物学功能的影响,结果显示PITPNB可以促进CNE-2细胞的增殖和迁移;而TargetScan分析显示,miR-1185-5p与PITPNB 3′UTR之间存在结合位点,推测miR-1185-5p与PITPNB之间可能存在靶向调控关系。本文应用双荧光素酶报告基因检测技术,首次在鼻咽癌CNE-2细胞中验证了miR-1185-5p与PITPNB之间的靶向结合关系。因此,PITPNB可能作为miR-1185-5p的下游靶点之一在鼻咽癌的发生发展中发挥作用。
综上,在鼻咽癌CNE-2细胞中,miR-1185-5p可通过靶向调控PITPNB的表达进而调节CNE-2细胞的增殖和迁移,从而抑制鼻咽癌的进展。这为鼻咽癌的治疗提供了新的靶标分子,为miR-1185-5p及PITPNB在鼻咽癌中作用的后续研究提供了生物学基础。然而,本研究仅局限于细胞实验,需动物实验进一步验证,并探索可能的上下游机制,为后续临床转化提供理论基础。
| [1] |
WEI W I, SHAM J S T. Nasopharyngeal carcinoma[J]. Lancet (London, England), 2005, 365(9476): 2041-2054. DOI:10.1016/S0140-6736(05)66698-6 |
| [2] |
CHUA M L K, WEE J T S, HUI E P, et al. Nasopharyngeal carcinoma[J]. Lancet (London, England), 2016, 387(10022): 1012-1024. DOI:10.1016/S0140-6736(15)00055-0 |
| [3] |
LU J C, TANG M, LI H D, et al. EBV-LMP1 suppresses the DNA damage response through DNA-PK/AMPK signaling to promote radioresistance in nasopharyngeal carcinoma[J]. Cancer Letters, 2016, 380(1): 191-200. DOI:10.1016/j.canlet.2016.05.032 |
| [4] |
KIM Y S, KIM B S, JUNG S L, et al. Radiation therapy combined with (or without) cisplatin-based chemotherapy for patients with nasopharyngeal cancer: 15-years experience of a single institution in Korea[J]. Cancer Research and Treatment, 2008, 40(4): 155-163. DOI:10.4143/crt.2008.40.4.155 |
| [5] |
CHEN L, HEIKKINEN L, WANG C L, et al. Trends in the development of miRNA bioinformatics tools[J]. Briefings in Bioinformatics, 2019, 20(5): 1836-1852. DOI:10.1093/bib/bby054 |
| [6] |
BARTEL D P. microRNAs[J]. Cell, 2004, 116(2): 281-297. DOI:10.1016/S0092-8674(04)00045-5 |
| [7] |
YUE W J, WANG Y, LI W Y, et al. LINC00887 regulates the proliferation of nasopharyngeal carcinoma via targeting miRNA-203b-3p to upregulate NUP205[J]. European Review for Medical and Pharmacological Sciences, 2020, 24(17): 8863-8870. |
| [8] |
TANG L, XIONG W, ZHANG L S, et al. circSETD3 regulates MAPRE1 through miR-615-5p and miR-1538 sponges to promote migration and invasion in nasopharyngeal carcinoma[J]. Oncogene, 2021, 40(2): 307-321. DOI:10.1038/s41388-020-01531-5 |
| [9] |
ZHENG Z Q, LI Z X, ZHOU G Q, et al. Long noncoding RNA FAM225A promotes nasopharyngeal carcinoma tumorigenesis and metastasis by acting as ceRNA to sponge miR-590-3p/miR-1275 and upregulate ITGB3[J]. Cancer Research, 2019, 79(18): 4612-4626. DOI:10.1158/0008-5472.CAN-19-0799 |
| [10] |
ZHU Q W, ZHANG Q C, GU M, et al. MIR106A-5p upregulation suppresses autophagy and accelerates malignant phenotype in nasopharyngeal carcinoma[J]. Autophagy, 2021, 17(7): 1667-1683. DOI:10.1080/15548627.2020.1781368 |
| [11] |
SHI Z H, WANG R S, HUANG L G, et al. Integrative analysis of miRNAs-mRNAs reveals that miR-182 up-regulation contributes to proliferation and invasion of nasopharyngeal carcinoma by targeting PTEN[J]. Aging, 2020, 12(12): 11568-11578. DOI:10.18632/aging.103316 |
| [12] |
XIAO Z F, CHEN Z C. Deciphering nasopharyngeal carcinoma pathogenesis via proteomics[J]. Expert Review of Proteomics, 2019, 16(6): 475-485. DOI:10.1080/14789450.2019.1615891 |
| [13] |
CAO S M, SIMONS M J, QIAN C N. The prevalence and prevention of nasopharyngeal carcinoma in China[J]. Chinese Journal of Cancer, 2011, 30(2): 114-119. DOI:10.5732/cjc.010.10377 |
| [14] |
CHAN A T C, GRÉGOIRE V, LEFEBVRE J L, et al. Nasopharyngeal cancer: EHNS-ESMO-ESTRO Clinical Practice Guidelines for diagnosis, treatment and follow-up[J]. Annals of Oncology: Official Journal of the European Society for Me- dical Oncology, 2010, 21(Suppl 5): v187-v189. |
| [15] |
LEE A W M, FOO W, LAW S C K, et al. Total biological effect on late reactive tissues following reirradiation for recurrent nasopharyngeal carcinoma[J]. International Journal of Radiation Oncology*Biology*Physics, 2000, 46(4): 865-872. DOI:10.1016/S0360-3016(99)00512-X |
| [16] |
HUANG T, YIN L, WU J, et al. microRNA-19b-3p regulates nasopharyngeal carcinoma radiosensitivity by targeting TNFAIP3/NF-κB axis[J]. Journal of Experimental & Clinical Cancer Research: CR, 2016, 35(1): 188. DOI:10.3760/cma.j.issn.1001-9030.2016.01.058 |
| [17] |
WANG S M, ZHANG R, CLARET F X, et al. Involvement of microRNA-24 and DNA methylation in resistance of nasopharyngeal carcinoma to ionizing radiation[J]. Molecular Cancer Therapeutics, 2014, 13(12): 3163-3174. DOI:10.1158/1535-7163.MCT-14-0317 |
| [18] |
FABIAN M R, SONENBERG N. The mechanics of miRNA-mediated gene silencing: a look under the hood of miRISC[J]. Nature Structural & Molecular Biology, 2012, 19(6): 586-593. |
| [19] |
DI LEVA G, GAROFALO M, CROCE C M. microRNAs in cancer[J]. Annual Review of Pathology, 2014, 9: 287-314. DOI:10.1146/annurev-pathol-012513-104715 |
| [20] |
CARSTENS J L, LOVISA S, KALLURI R. Microenvironment-dependent cues trigger miRNA-regulated feedback loop to facilitate the EMT/MET switch[J]. The Journal of Clinical Investigation, 2014, 124(4): 1458-1460. DOI:10.1172/JCI75239 |
| [21] |
USUBA W, URABE F, YAMAMOTO Y, et al. Circulating miRNA panels for specific and early detection in bladder cancer[J]. Cancer Science, 2019, 110(1): 408-419. DOI:10.1111/cas.13856 |
| [22] |
PORKKA K P, PFEIFFER M J, WALTERING K K, et al. microRNA expression profiling in prostate cancer[J]. Cancer Research, 2007, 67(13): 6130-6135. DOI:10.1158/0008-5472.CAN-07-0533 |
| [23] |
AKAO Y, NAKAGAWA Y, NAOE T. microRNA-143 and -145 in colon cancer[J]. DNA and Cell Biology, 2007, 26(5): 311-320. DOI:10.1089/dna.2006.0550 |
| [24] |
BRUCE J P, LIU F F. microRNAs in nasopharyngeal carcinoma[J]. Chinese Journal of Cancer, 2014, 33(11): 539-544. DOI:10.5732/cjc.014.10175 |
| [25] |
LIU B, LI J Y, CAIRNS M J. Identifying miRNAs, targets and functions[J]. Briefings in Bioinformatics, 2012, 15(1): 1-19. |
| [26] |
ALQURASHI N, HASHIMI S M, ALOWAIDI F, et al. miR-496, miR-1185, miR-654, miR-3183 and miR-495 are downregulated in colorectal cancer cells and have putative roles in the mTOR pathway[J]. Oncology Letters, 2019, 18(2): 1657-1668. |
| [27] |
XU Y Q, CHEN W C, LIANG J, et al. The miR-1185-2-3p-GOLPH3L pathway promotes glucose metabolism in breast cancer by stabilizing p53-induced SERPINE1[J]. Journal of Experimental & Clinical Cancer Research: CR, 2021, 40(1): 47. |
 2022, Vol. 58
2022, Vol. 58

