2. 青岛市疾病预防控制中心, 山东 青岛 266001
马凡综合征(MFS)是最常见的常染色体显性遗传性结缔组织疾病之一[1], 其发病机制不明[2]。目前, 国内相关研究报道MFS发病率为0.04%~0.10%[3], 其临床表现主要累及心血管系统、骨骼系统、眼和神经系统等[4]。该疾病一旦累及心血管系统[5], 则病人极有可能死于心脏衰竭或主动脉瘤破裂[6-7]。为更深入了解MFS遗传学特征, 本研究通过利用专业数据库查阅相关文献, 尝试分析某些药物以及基因/蛋白引起MFS的可能性及其可能机制。现将结果报告如下。
1 资料和方法 1.1 基因筛选以MFS(MESH:D008382)作为目标疾病, 应用比较毒理基因组学数据库(CTD)[8]与STRING数据库筛选与MFS关联的药物及基因的数据集。
1.2 通路和生物进程功能富集分析分别利用http://string-db.org/中的string[9]和http://metascape.org中的metascape[10]对关键基因进行注释和进一步分析。参考的数据资源包括KEGG通路、GO生物学进程、Reactome基因组、经典通路和哺乳动物蛋白复合物数据库(CORUM)。根据相关文献方法[11-13]进行通路和生物进程功能富集分析。
1.3 蛋白-蛋白互作富集分析应用Metascape BioGrid[14]、InWeb_IM以及OmniPath[15]数据库对蛋白-蛋白互作进行富集分析。应用MCODE算法[16]对互作蛋白进行模块化分析, 每个模块的蛋白进一步进行通路和生物进程富集分析。根据P值确定最佳前3位的通路或生物进程作为所在模块的功能描述。
2 结果 2.1 与MFS相关的基因/蛋白以MFS作为目标疾病, 通过CTD数据库检索显示, 有258种基因与MFS有关, 其中基因评价分值≥5.5有26种基因, 见表 1。其中GJA1基因与MFS联系最为密切, 其次为ABCB11基因。
| 表 1 与MFS相关的常见基因/蛋白 |
|
|
CTD数据库检索显示, 1 546种药物与MFS有关。其中基因评价分值≥5.5的26种基因对应的药物见表 2, 有阿替洛尔、多四环素、氯沙坦、硫代巴比妥酸等4种药物与26种基因密切相关。其中氯沙坦与26种基因相关程度达84.62%。
| 表 2 26种基因/蛋白对应药物 |
|
|
STRING数据库分析结果显示, 有24种基因在STRING数据库识别(除SNORA49、PINK-1)(图 1)。24种基因之间存在着密不可分的互作关系, 基因与基因、蛋白与蛋白的相互作用可以通过基因数据库和实验来验证, 还可能通过基因间相邻作用、基因间相互结合与基因间相互同现而发生相互联系, 一些基因转录文本、基因间共表达及基因同源性都可以导致基因与基因、蛋白与蛋白相互作用。
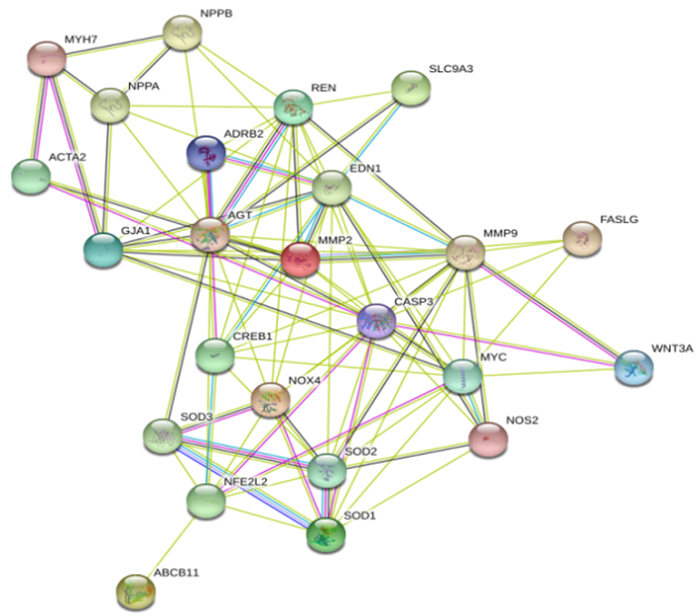 |
| NHE3=SLC9A3。 图 1 STRING数据库识别与人类相关的24种基因图 |
应用metascape进一步对24种关键基因富集分析, 将富集后的结果按功能分簇, 结果见表 3。24种关键基因主要集中在循环系统、超氧化物代谢过程、凋亡过程的正向调节、血压的负向调节、细胞对有机氮的反应、转化生长因子β信号、多细胞的生物过程、肾脏发育、脂多糖反应、钠离子运输过程以及有机羟基化合物转运等GO生物学进程, 与蛋白多糖反应、乙型肝炎、肾素分泌以及激活丝裂原活化蛋白激酶信号通路等信号通路相关。
| 表 3 24种关键基因/蛋白富集分析结果 |
|
|
应用Cytoscape中MCODE模块化分析结果显示, 24种基因/蛋白共有2个基因模块(MCODE1和MCODE2)(图 2)。NOS2、REN、AGT、MMP9、MYC、CREB1、EDN1与ACTA2、CASP3、MMP2分别通过结合、同现、共表达以及同源性相互作用。MCODE1中7种基因/蛋白(NOS2、REN、AGT、CREB1、EDN1、MMP9、MYC)参与相关生物过程, 包括转化生长因子β信号生物过程(CREB1、MYC以及EDN1)、激活丝裂原活化蛋白激酶信号通路(AGT、CREB1、MYC、EDN1、REN)、NO正向调节(AGT、EDN1、NOS2)及纤维蛋白原正向调节通路(AGT、MYC); 而模块MCODE2中3种基因/蛋白(ACTA2、CASP3、MMP2)不参与相关生物过程。
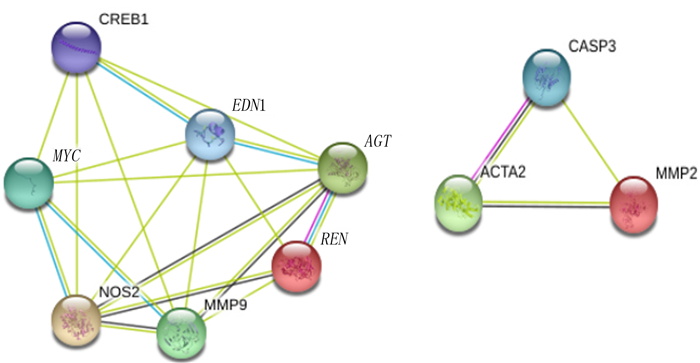 |
| MCODE1:7种基因/蛋白形成的模型簇; MCODE2:3种基因/蛋白形成的模型簇。 图 2 两个模型簇图 |
本文通过对MFS与人类相关的24种主要基因/蛋白富集显示, MFS相关基因/蛋白主要集中在循环系统、超氧化物代谢过程、凋亡过程的正向调节、血压的负向调节、细胞对有机氮的反应、转化生长因子β信号、多细胞的生物过程、肾脏发育、脂多糖反应、钠离子运输过程以及有机羟基化合物转运、蛋白多糖反应、乙型肝炎、肾素分泌以及激活丝裂原活化蛋白激酶信号通路等信号通路。其中转化生长因子超家族信号通路对维持细胞外基质和组织稳态起着重要的作用[17-18]。
国内外相关学者研究认为, 原纤维蛋白-1基因(FBN1)是MFS致病的主要基因[19-21]。FBN1是一种抗胶原蛋白酶的非硫酸化的糖蛋白, 参与蛋白多糖反应生物过程, 为细胞外基质微纤维的重要组成成分[22]; 同时还可以调节转化生长因子β(TGF-β)的释放[23-25]。有研究表明, FBN1突变会引起结构改变, 不仅可以导致弹性纤维组装障碍, 而且还会引起TGF-β的过度激活, 从而引起TGF-β介导的信号传导增强[26]。KIELTY等[27]研究发现, 由于基因突变引起的微纤维蛋白异常可能导致血管弹性纤维改变, 从而使病人更容易发生主动脉夹层或形成主动脉瘤[28-29]。
TGF-β的过度激活与细胞外基质中MMPs的异常表达有关, MMPs作为一种锌内肽酶, 其中的MMP2和MMP9是经典TGF-β通路的下游调控产物[30]。CHUNG等[31]研究显示, TGF-β针对性诱导MMP2和MMP9高表达, 在实验诱导以及自然形成的动脉瘤组织中都可以观察到MMPs高表达[24], 而MMPs高表达使得主动脉相关蛋白质分解从而诱导动脉瘤形成[32-33]。DIETZ[34]对MFS小鼠模型研究发现, 主动脉壁组织中确实存在TGF-β信号转导通路过度激活的现象, 使用TGF-β信号转导通路阻断剂氯沙坦, 可阻断MFS小鼠主动脉病变的进展; 同样, FORTEZA等[7]研究显示, 血管紧张素Ⅱ受体阻滞剂氯沙坦能抑制TGF-β介导的细胞外信号调节激酶激活, 进而延缓甚至逆转主动脉扩张等病变的进展, 从而为MFS血管病变的治疗提供有力临床证据[37]。本文通过CTD数据库初步分析显示, 氯沙坦与26种基因密切相关, 因此, 氯沙坦可应用于治疗MFS病人。
本文通过基因/蛋白富集分析显示, TGF-β还可激活其他信号传导通路, 该通路主要为丝裂原活化的蛋白激酶(MAPK)信号转导通路, 涉及AGT、CREB1、MYC、EDN1、REN等相关基因, 这与国内外报道的结果相符[36-39]。这些基因通过结合、同现、共表达以及同源性相互作用共同作用于MAPK信号转导通路[40], 该通路上还有细胞外信号调节激酶(ERK)、Jun氨基端激酶(JNK)和P38。在MFS血管组织病变过程中主要是ERK起决定作用[41], 这可能为治疗MFS血管病变提供新的靶点。
同时我们的研究结果也显示, 环境因素一氧化氮(NO)也可能对MFS血管病变有一定的影响。这与CHUNG等[42]应用MFS小鼠模型的研究结果相似, MFS与内皮NO合成减少有关, 并推测血管内皮功能紊乱可能是MFS病人主动脉并发症发生机制之一。
综上所述, MFS的发病机制目前尚不明确, 而国内外的研究多集中在实验动物模型, 但仅凭动物研究结果并不能说明其在人类中的机制。本文借助CTD数据库和其他在线分析工具研究显示, 药物可能通过影响基因表达从而引起某些信号通路的改变等内在分子生物学机制与MFS产生关联, 而该种关联尚需要在人群中进一步验证。
| [1] |
武玉敏, 崔建, 阮银镜, 等. 一家四代马凡综合征6例报告[J]. 实用医学杂志, 2005, 21(9): 999. |
| [2] |
冯彤. 马凡氏综合征心血管病变的临床与影像学表现[J]. 影像研究与医学应用, 2018, 2(4): 64-65. |
| [3] |
那开宪. 马凡氏综合征[J]. 首都食品与医药, 2017(15): 58-59. |
| [4] |
贺晶, 高凌根. 马凡综合征的分子遗传学研究进展[J]. 中华保健医学杂志, 2015, 17(5): 421-424. |
| [5] |
苏育琳, 陈子怡. 马凡氏综合征继发脑血管疾病的临床分析[J]. 中国神经精神疾病杂志, 2019, 45(1): 47-50. |
| [6] |
覃勇民, 伍广伟. 马凡综合征的临床研究进展[J]. 疑难病杂志, 2010, 9(10): 797-801. |
| [7] |
FORTEZA A, EVANGELISTA A, SÁNCHEZ V, et al. Efficacy of losartan vs atenolol for the prevention of aortic dilation in Marfan Syndrome: a randomized clinical trial[J]. European Heart Journal, 2015, 37(12): 978-985. |
| [8] |
DAVIS A P, GRONDIN C J, JOHNSON R J, et al. The comparative toxicogenomics database: update 2019[J]. Nucleic Acids Research, 2019, 47(D1): D948-D954. |
| [9] |
SZKLARCZYK D, MORRIS J H, COOK H, et al. The STRING database in 2017: quality-controlled protein-protein association networks, made broadly accessible[J]. Nucleic Acids Research, 2017, 45(D1): D362-D368. |
| [10] |
ZHOU YY, ZHOU B, PACHE L, et al. Metascape provides a biologist-oriented resource for the analysis of systems-level datasets[J]. Nature Communications, 2019, 10(1): 1523-1531. |
| [11] |
JERROLD Z H. Biostatistical analysis[M]. India: Pearson Education India, 1999.
|
| [12] |
HOCHBERG Y, BENJAMINI Y. More powerful procedures for multiple significance testing[J]. Statistics in Medicine, 1990, 9(7): 811-818. |
| [13] |
COHEN J. A coefficient of agreement for nominal scales[J]. Educational and Psychological Measurement, 1960, 20(1): 37-46. |
| [14] |
STARK C, BREITKREUTZ B J, REGULY T, et al. BioGRID: a general repository for interaction datasets[J]. Nucleic Acids Research, 2006, 34(Database issue): D535-D539. |
| [15] |
LI T B, WERNERSSON R, HANSEN R B, et al. A scored human protein-protein interaction network to catalyze genomic interpretation[J]. Nature Methods, 2017, 14(1): 61-64. |
| [16] |
BADER G D, HOGUE C W. An automated method for finding molecular complexes in large protein interaction networks[J]. BMC Bioinformatics, 2003, 4(1): 2-10. |
| [17] |
BERTOLI-AVELLA A M, GILLIS E M, MORISAKI H, et al. Mutations in a TGFβ ligand, TGFB3, cause syndromic aortic aneurysms and dissections[J]. Journal of the American College of Cardiology, 2015, 65(3): 1324-1336. |
| [18] |
PEPE G, GIUSTI B, STICCHI E, et al. Marfan syndrome:current perspectives[J]. Appl Clin Genet, 2016, 9: 55-65. |
| [19] |
VERSTRAETEN A, ALAERTS M, VAN LAER L, et al. Marfan syndrome and related disorders:25 years of gene discovery[J]. Human Mutation, 2016, 37(6): 524-531. |
| [20] |
张琳, 张莹, 范鹏, 等. FBN1基因钙结合转化生长因子结构域终止突变基因型表型研究[J]. 中国分子心脏病学杂志, 2018, 18(3): 2513-2516. |
| [21] |
FRANKEN R, GROENINK M, DE WAARD V, et al. Genotype impacts survival in Marfan syndrome[J]. European Heart Journal, 2015, 36(1): 523-527. |
| [22] |
GUGLIELMINA P, GIUSTI B, STICCHI E, et al. Marfan syndrome:current perspectives[J]. The Application of Clinical Genetics, 2016, 9: 55-65. |
| [23] |
张琳, 张莹, 范鹏, 等. 2型马凡综合征TGFBR2基因新致病突变研究[J]. 中国心血管杂志, 2018, 23(4): 288-293. |
| [24] |
公兵, 杨秀滨. 马凡氏综合征血管病变的基础研究进展[J]. 心脏杂志, 2012, 24(6): 760-762. |
| [25] |
CANNAERTS E, BERTOLI-AVELLA A, GILLIS E, et al. Mutations in a TGFβ ligand, TGFB3, cause syndromic aortic aneurysms and dissections[J]. Journal of the American College of Cardiology, 2015, 65(13): 1324-1336. |
| [26] |
SENGLE G, TSUTSUI K, KEENE D R, et al. Microenvironmental regulation by fibrillin-1[J]. PLoS Genetics, 2012, 8(1): e1002425. |
| [27] |
KIELTY C M, SHERRATT M J, MARSON A, et al. Fibrillin microfibrils[J]. Advances in Protein Chemistry, 2005, 70: 405-436. |
| [28] |
FAIVRE L, MASUREL-PAULET A, COLLOD-BEROUD G, et al. Clinical and molecular study of 320 children with marfan syndrome and related type Ⅰ fibrillinopathies in a series of 1009 probands with pathogenic FBN1 mutations[J]. Pediatrics, 2009, 123(1): 391-398. |
| [29] |
LI-WAN-PO A, LOEYS B, FARNDON P, et al. Preventing the aortic complications of Marfan syndrome: a case-example of translational genomic medicine[J]. British Journal of Clinical Pharmacology, 2011, 72(1): 6-17. |
| [30] |
刘鼎乾, 李军, 赖颢. TGF-β信号通路与马方综合征及Loeyz-Dietz综合征研究进展[J]. 中国分子心脏病学杂志, 2018, 18(1): 2386-2389. |
| [31] |
CHUNG A W, YANG H H, RADOMSKI M W, et al. Long-term doxycycline is more effective than atenolol to prevent thoracic aortic aneurysm in marfan syndrome through the inhibition of matrix metalloproteinase-2 and -9[J]. Circulation Research, 2008, 102(8): e73-e85. |
| [32] |
WU D, SHEN Y H, RUSSELL L, et al. Molecular mechanisms of thoracic aortic dissection[J]. Journal of Surgical Research, 2013, 184(2): 907-924. |
| [33] |
FRANKEN R, DEN HARTOG A W, RADONIC T, et al. Beneficial outcome of losartan therapy depends on type of FBN1 mutation in marfan syndrome[J]. Circulation-Cardiovascular Genetics, 2015, 8(2): 383-388. |
| [34] |
DIETZ H C. TGF-beta in the pathogenesis and prevention of disease: a matter of aneurysmic proportions[J]. The Journal of Clinical Investigation, 2010, 120(2): 403-407. |
| [35] |
BROOKE B S, HABASHI J P, JUDGE D P, et al. Angiotensin Ⅱ blockade and aortic-root dilation in Marfan's syndrome[J]. New England Journal of Medicine, 2008, 358(26): 2787-2795. |
| [36] |
LOEYS B, DE PAEPE A. New insights in the pathogenesis of aortic aneurysms[J]. Verhandelingen-Koninklijke Academie voor Geneeskunde Van Belgie, 2008, 70(2): 69-84. |
| [37] |
NG C M, CHENG A L, MYERS L A, et al. TGF-beta-dependent pathogenesis of mitral valve prolapse in a mouse model of Marfan syndrome[J]. The Journal of Clinical Investigation, 2004, 114(11): 1586-1592. |
| [38] |
HABASHI J P, DOYLE J J, HOLM T M, et al. Angiotensin Ⅱ type 2 receptor signaling attenuates aortic aneurysm in mice through ERK antagonism[J]. Science (New York, N.Y.), 2011, 332(627): 361-365. |
| [39] |
LEMAIRE R, FARINA G, BAYLE J, et al. Antagonistic effect of the matricellular signaling protein CCN3 on TGF-beta- and Wnt-mediated fibrillinogenesis in systemic sclerosis and Marfan syndrome[J]. The Journal of Investigative Dermatology, 2010, 130(6): 1514-1523. |
| [40] |
LIN C Y, CHIN C H, WU H H, et al. Hubba: hub objects analyzer-a framework of interactome hubs identification for network biology[J]. Nucleic Acids Research, 2008, 36(S): W438-W443. |
| [41] |
FRANKEN R, DEN HARTOG A W, DE WAARD V, et al. Circulating transforming growth factor-β as a prognostic biomarker in Marfan syndrome[J]. International Journal of Cardiology, 2013, 168(3): 2441-2446. |
| [42] |
CHUNG A W, AU YEUNG K, CORTES S F, et al. Endothelial dysfunction and compromised eNOS/Akt signaling in the thoracic aorta during the progression of Marfan syndrome[J]. British Journal of Pharmacology, 2007, 150(8): 1075-1083. |
 2020, Vol. 56
2020, Vol. 56

