全球女性中乳癌的发病率和死亡率占女性恶性肿瘤之首[1]。目前,治疗乳癌的主要方法包括手术治疗、放疗和化疗等,但有侵入性强、副作用明显等缺点,对病人身体和心理上有一定程度的损伤[2-4]。因此,寻找新的治疗手段非常重要。DNA聚合酶β(DNA pol β)基因已被证实与多种肿瘤的发生、发展有密切的关系[5-6]。在正常情况下,DNA polβ的表达保持着持续的低水平,而在肿瘤细胞中高表达,并常伴随着结构突变[7]。然而,DNA polβ基因在乳癌细胞中作用研究则少报道。在本文中,我们研究DNA polβ基因对乳癌细胞的增殖和侵袭的影响,探究其是否可作为治疗乳癌的新靶点。
1 材料与方法 1.1 材料人乳癌细胞MCF-7购买于中桥新舟;转染试剂脂质体2000购自Invitrogen公司;总RNA提取试剂盒购于北京百泰克生物技术有限公司;反转录试剂Quantscript RT Kit与荧光定量试剂SuperReal、荧光定量预混试剂彩色版(SYBR Green)购于天根生化科技(北京)有限公司;BCA蛋白浓度测定试剂盒购于碧云天生物技术有限公司;抗DNA polβ和β-actin抗体购于Santa Cruz生物公司;辣根过氧化物酶标记山羊抗小鼠IgG(H+L)购于碧云天生物技术有限公司;脂质体2000购于Invitrogen公司;噻唑蓝(MTT)与二甲基亚砜(DMSO)购于Sigma公司;Matrigel胶购于BD公司。本实验中所用引物由上海生工生物工程有限公司合成,上游引物序列为CCGCAGGAGACTCTCAACG,下游引物序列为GTACTTGTGGATAGCTTGGCTC。
1.2 方法 1.2.1 质粒构建与转染构建3种Pslience 2.0-DNA polβ siRNA质粒和1种Pslience 2.0-NC-siRNA对照质粒。以不加质粒的MCF-7细胞为阴性Control组(C组)、以NC-siRNA为对照质粒组(D组)、以1~3号DNA polβ-siRNA质粒为敲降组(E组、F组、G组),筛选DNA polβ-siRNA的敲降效率。培养MCF-7细胞融合达到85%左右,将细胞消化后接种于6孔板中。利用脂质体2000将对照质粒、DNA polβ质粒分别转染进入MCF-7细胞,37 ℃恒温培养24 h后检测转染效率。选择敲降效率最好的siRNA质粒进行后续实验,分组为Control组(A组)、NC-siRNA组(B组)和DNA polβ-siRNA组(C组)。
1.2.2 荧光定量PCR(RT-PCR)收集细胞,以总RNA提取试剂盒提取总RNA,使用反转录试剂盒反转录总RNA得到样品cDNA。RT-PCR实验检测各组细胞中DNA polβ基因的表达水平。本实验采用20 μL反应体系:cDNA模板1 μL,上下游引物各0.5 μL,SYBR GREEN master mix 10 μL,ddH2O补齐至20 μL。实验重复3次,结果分析采用2-△△CT方法[8]。
1.2.3 蛋白质印迹(Western blot)实验RIPA裂解液裂解细胞后,以BCA蛋白浓度测定试剂盒检测蛋白浓度。同质量蛋白质样品经聚丙烯酰胺凝胶电泳进行分离。将蛋白转印至PVDF膜上,50 g/L脱脂牛奶孵育PVDF膜1 h。利用DNA polβ抗体和β-actin抗体分别孵育PVDF膜,4 ℃过夜。一抗孵育后,使用辣根过氧化物酶标记的山羊抗小鼠IgG(H+L)作为二抗,孵育PVDF膜(37 ℃、45 min)。二抗孵育后,使用ECL发光液处理,并在暗室中拍照。β-actin作为内参照。
1.2.4 MTT检测MCF-7细胞转染质粒后培养48 h,加入浓度为5 g/L的MTT溶液,放置于37 ℃恒温培养箱继续培养4 h。取出细胞,吸除上清,向细胞中加入200 μL的DMSO溶液。利用酶标仪测定各组细胞在490 nm波长处的吸光度(A)值,并进行数据分析。
1.2.5 Transwell实验用Matrigel胶包被Transwell小室,放置于37 ℃培养箱中2 h。将Transwell小室放置于24孔板上,下室加入含体积分数0.30胎牛血清的培养液800 μL,上室加入200 μL、2.5 g/L胰蛋白酶消化后的单细胞悬液,细胞数均为每孔1×104个。将24孔板置于37 ℃、含体积分数0.05 CO2、饱和湿度条件下的细胞培养箱中培养24 h。培养后,Transwell小室使用40 g/L多聚甲醛室温下固定20 min,5 g/L甲紫染液染色5 min。使用倒置光学显微镜(200倍)对迁移微孔膜下层的细胞进行计数。
1.3 统计学方法本实验数据统计分析和作图均使用SPSS 13.0软件。计量资料数据用x±s表示,组间比较采用单因素方差分析中Bonferroni方法进行数据分析。以P < 0.05为差异具有显著性。
2 结果 2.1 DNA polβ-siRNA敲降效率本实验利用RT-PCR和Western blot技术检测DNA polβ-siRNA的敲降效率,结果表明,3种DNA polβ-siRNA转染MCF-7细胞后,DNA polβ的mRNA和蛋白水平均明显低于转染NC-siRNA组(F=8.6,P < 0.05)。见图 1。在敲降MCF-7细胞中以DNA polβ-siRNA-2质粒的效率最高,采用其进行后续实验。
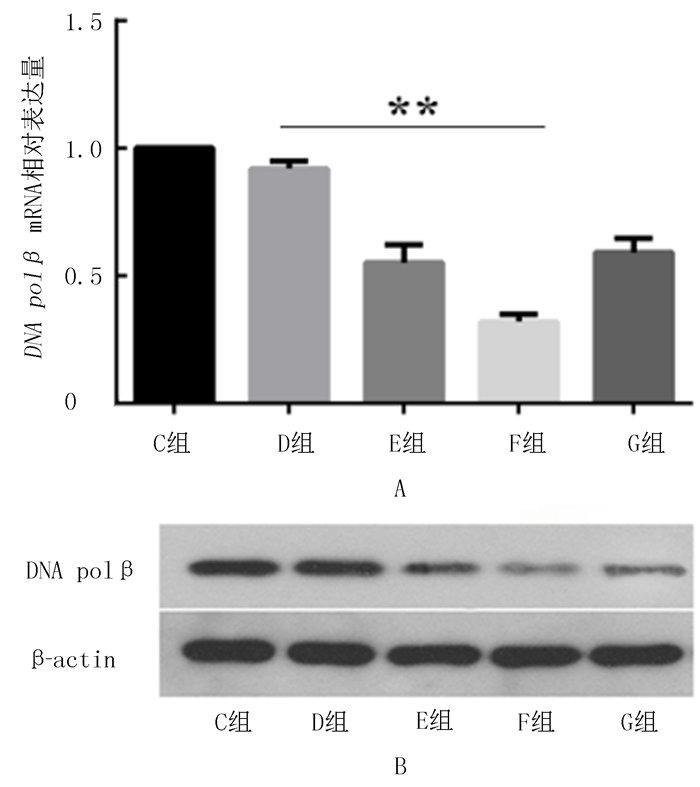 |
| C组:Control,D组:NC-siRNA,E组:DNA polβ-siRNA-1,F组:DNA polβ-siRNA-2,G组:DNA polβ-siRNA-3。与D组相比,E组、F组、G组MCF-7细胞中DNA polβ表达显著降低。 图 1 RT-PCR和Western blot检测 |
MTT检测结果表明,MCF-7细胞转染DNA polβ-siRNA质粒后,DNA polβ-siRNA转染组细胞增殖明显低于NC-siRNA转染组,两组比较差异具有显著性(F=5.8,P < 0.05)。见图 2。
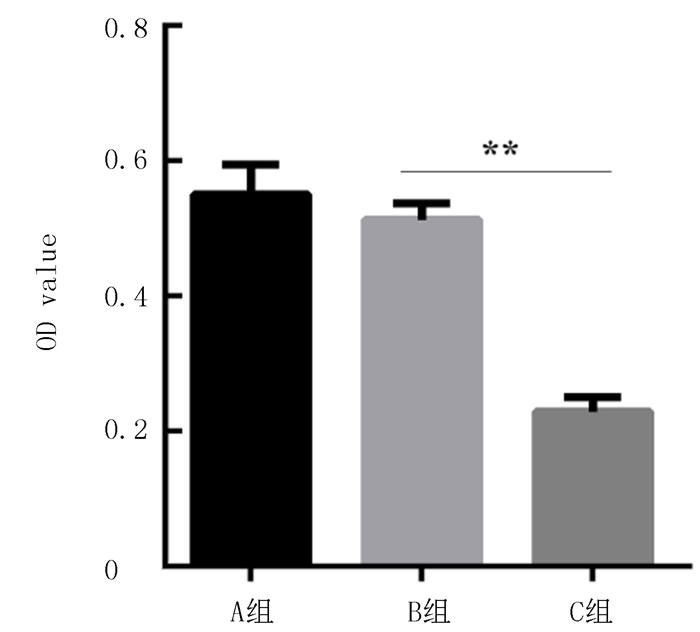 |
| A组:Control组,B组:NC-siRNA组,C组:DNA polβ-siRNA组。DNA polβ-siRNA显著抑制MCF-7细胞增殖。 图 2 MTT检测DNA polβ-siRNA对细胞增殖影响 |
Transwell实验的检测结果显示,乳癌MCF-7细胞DNA polβ敲降后,DNA polβ-siRNA转染组细胞的侵袭能力明显低于NC-siRNA转染组,两组相比较差异具有统计学意义(F=5.2,P < 0.05)。见图 3。
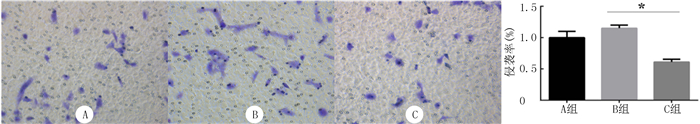 |
| A:Control组,B:NC-siRNA组,C:DNA polβ-siRNA组。DNA polβ-siRNA可显著抑制MCF-7细胞侵袭能力。 图 3 Transwell实验检测DNA polβ-siRNA对细胞侵袭影响 |
乳癌在多个国家中是女性发病率最高的恶性肿瘤,严重影响女性的身心健康,受到广泛关注[9-11]。随着分子技术的不断发展,利用基因治疗手段治疗乳癌受到广泛关注,其具有更精准、快速且无副作用的优势[12-14]。
DNA polβ是DNA聚合酶家族中的重要成员,在DNA碱基切除修复中起到重要作用[15],该酶与肿瘤的形成密切相关[16-19]。DNA polβ基因已被证实与食管癌[20-21]、胃癌[22-23]、眼睑基底细胞癌[24]、宫颈癌[25]、乳癌[26-27]等多种癌症的发生具有密切关系。研究结果表明,DNA polβ基因在乳癌组织中的表达明显高于癌旁组织,在正常的人乳腺上皮细胞MCF-10A中DNA polβ基因的表达也明显低于人乳癌上皮细胞株MCF-7[28-29],提示DNA polβ基因的表达上调,有可能是导致乳癌恶性增生的机制之一[30]。因此,有效干预DNA polβ过表达,可能对于乳癌的有效防治具有重要理论和实用意义。
本研究采用RNA干扰技术抑制MCF-7细胞中的DNA polβ的表达。结果显示,在抑制DNA polβ的表达后,MCF-7细胞的增殖和侵袭能力均受到显著抑制,与文献报道结果相一致。说明DNA polβ可能是参与乳癌发展过程中重要的因素之一。
综上所述,DNA polβ基因有可能作为治疗乳癌的关键基因靶点,本文结果为乳癌临床治疗的选择提供新的理论依据。但是本文具有一定的局限性,DNA polβ在治疗乳癌中的应用还需要更多的理论研究与临床试验证明。
| [1] |
TORRE L A, ISLAMI F, SIEGEL R L, et al. Global cancer in women:burden and trends[J]. Cancer Epidemiology Biomarkers & Prevention, 2017, 26(4): 444-457. |
| [2] |
Early Breast Cancer Trialists' Collaborative Group. Effects of chemotherapy and hormonal therapy for early breast cancer on recurrence and 15-year survival:an overview of the randomised trials[J]. The Lancet, 2005, 365(9472): 1687-1717. DOI:10.1016/S0140-6736(05)66544-0 |
| [3] |
FISHER B, BRYANT J, WOLMARK N, et al. Effect of preoperative chemotherapy on the outcome of women with operable breast cancer[J]. Journal of Clinical Oncology, 1998, 16(8): 2672-2685. DOI:10.1200/JCO.1998.16.8.2672 |
| [4] |
FISHER B, ANDERSON S, BRYANT J, et al. Twenty-year follow-up of a randomized trial comparing total mastectomy, lumpectomy, and lumpectomy plus irradiation for the treatment of invasive breast cancer[J]. The New England Journal of Medicine, 2002, 347(16): 1233-1241. DOI:10.1056/NEJMoa022152 |
| [5] |
BRAY F, JEMAL A, GREY N, et al. Global cancer transitions according to the Human Development Index (2008-2030):a population-based study[J]. The Lancet Oncoloy, 2012, 13(8): 790-801. DOI:10.1016/S1470-2045(12)70211-5 |
| [6] |
WANG Yuanyuan, ZANG Wenqiao, DU Yuwen, et al. The K167I variant of DNA polymerase beta that is found in Esophageal carcinoma patients impairs polymerase activity and BER[J]. Scientific Reports, 2015, 5: 15986. DOI:10.1038/srep15986 |
| [7] |
LI Min, ZANG Wenqiao, WANG Yuanyuan, et al. DNA polymerase beta promoter mutations and transcriptional activity in esophageal squamous cell carcinoma[J]. Tumour Biology, 2013, 34(5): 3259-3263. DOI:10.1007/s13277-013-0898-5 |
| [8] |
LIVAK K J, SCHMITTGEN D T. Analysis of relative gene expression data using real-time quantitative PCR and the 2(-Delta Delta C(T)) method[J]. Methods, 2001, 25(4): 402-408. DOI:10.1006/meth.2001.1262 |
| [9] |
ANASTASIADI Z, LIANOS G D, IGNATIADOU E, et al. Breast cancer in young women:an overview[J]. Updates in Surgery, 2017, 69(3): 313-317. DOI:10.1007/s13304-017-0424-1 |
| [10] |
WOOLSTON C. Breast cancer[J]. Nature, 2015, 527(7578): S101. DOI:10.1038/527S101a |
| [11] |
MERINO BONILLA J A, TORRES TABANERA M, ROS MENDOZA L H. Breast cancer in the 21st century:from early detection to new therapies[J]. Radiologia, 2017, 59(5): 368-379. DOI:10.1016/j.rx.2017.06.003 |
| [12] |
BRADEN Y M, STANKOWSKI R V, ENGEL J M, et al. Breast cancer biomarkers:risk assessment, diagnosis, prognosis, prediction of treatment efficacy and toxicity, and recurrence[J]. Current Pharmaceutical Design, 2014, 20(30): 4879-4898. DOI:10.2174/1381612819666131125145517 |
| [13] |
VAN'T VEER L J, DAI H, VAN DE VIJVER M J, et al. Gene expression profiling predicts clinical outcome of breast cancer[J]. Nature, 2002, 415(6871): 530-536. DOI:10.1038/415530a |
| [14] |
CAMPEAU P M, FOULKES W D, TISCHKOWITZ M D. Hereditary breast cancer:new genetic developments, new therapeutic avenues[J]. Human Genetics, 2008, 124(1): 31-42. |
| [15] |
SYKORA P, KANNO S, AKBARI M, et al. DNA Polymerase Beta participates in mitochondrial DNA repair[J]. Molecular & Cellular Biology, 2017, 37(16): e00237-00217. |
| [16] |
BERGOGLIO V, PILLAIRE M J, LACROIX-TRIKI M, et al. Deregulated DNA polymerase beta induces chromosome instability and tumorigenesis[J]. Cancer Research, 2002, 62(12): 3511-3514. |
| [17] |
BALUSU R, JAISWAL A S, ARMAS M L, et al. Structure/function analysis of the interaction of adenomatous polyposis coli with DNA polymerase beta and its implications for base excision repair[J]. Biochemistry, 2007, 46(49): 13961-13974. DOI:10.1021/bi701632e |
| [18] |
KHANRA K, CHOUDHURI I, N B. Detection of somatic mutation in exon 12 of DNA polymerase beta in ovarian cancer tissue samples[J]. Iranian Biomedical Journal, 2018, 22(5): 355-359. DOI:10.29252/ibj.22.5.355 |
| [19] |
NEIJENHUIS S, VERWIJS-JANSSEN M, VAN DEN BROEK L J, et al. Targeted radiosensitization of cells expressing truncated DNA polymerase beta[J]. Cancer Resreach, 2010, 70(21): 8706-8714. DOI:10.1158/0008-5472.CAN-09-3901 |
| [20] |
乔玉环, 韩丽萍, 史惠蓉. 宫颈癌组织中DNA聚合酶β基因突变研究[J]. 肿瘤防治杂志, 2002, 9(5): 482-484. DOI:10.3969/j.issn.1673-5269.2002.05.014 |
| [21] |
WANG Yuanyuan, CHEN Xiaonan, SUN Qianqian, et al. Overexpression of A613T and G462T variants of DNA polymerase beta weakens chemotherapy sensitivity in esophageal cancer cell lines[J]. Cancer Cell International, 2016, 16(1): 85. DOI:10.1186/s12935-016-0362-x |
| [22] |
IWANAGA A, OUCHIDA M, MIYAZAKI K, et al. Functional mutation of DNA polymerase beta found in human gastric cancer-inability of the base excision repair in vitro[J]. Mutation Research, 1999, 435(2): 121-128. DOI:10.1016/S0921-8777(99)00036-1 |
| [23] |
SIMONELLI V, LEUZZI G, BASILE G, et al. Crosstalk between mismatch repair and base excision repair in human gastric cancer[J]. Oncotarget, 2017, 8(49): 84827. |
| [24] |
张千帆, 彭波. DNA聚合酶β基因在眼睑基底细胞癌中的表达及意义[J]. 河南大学学报(医学版), 2014, 33(4): 274-276. |
| [25] |
段娥, 李英勇. DNA聚合酶β在宫颈上皮内瘤变和宫颈癌中的表达及临床意义[J]. 国际医药卫生导报, 2015, 21(1): 9-11. DOI:10.3760/cma.j.issn.1007-1245.2015.01.003 |
| [26] |
丁继强, 宁召锋, 李梁, 等. DNA聚合酶β高表达与食管癌放化疗敏感性的临床研究[J]. 肿瘤基础与临床, 2016, 29(3): 201-205. DOI:10.3969/j.issn.1673-5412.2016.03.006 |
| [27] |
ALI R, RAKHA E A, MADHUSUDAN S, et al. DNA damage repair in breast cancer and its therapeutic implications[J]. Pathology, 2017, 49(2): 156-165. DOI:10.1016/j.pathol.2016.11.002 |
| [28] |
郑英斌, 韩娜, 侯帅恒, 等. 乳腺癌中DNA聚合酶-β的表达及其对乳腺癌细胞迁移的影响[J]. 中华实验外科杂志, 2016, 33(9): 2135-2137. DOI:10.3760/cma.j.issn.1001-9030.2016.09.022 |
| [29] |
郑英斌, 赵文超, 解璞, 等. 乳腺癌中DNA聚合酶-β的表达及其对乳腺癌细胞增殖的影响[J]. 中华实验外科杂志, 2016, 33(10): 2386-2388. DOI:10.3760/cma.j.issn.1001-9030.2016.10.042 |
| [30] |
DONG Z M, ZHENG N G, WU J L, et al. Difference in expression level and localization of DNA polymerase beta among human esophageal cancer focus, adjacent and corresponding normal tissues[J]. Diseases of the Esophagus, 2006, 19(3): 172-176. DOI:10.1111/j.1442-2050.2006.00560.x |
 2019, Vol. 55
2019, Vol. 55

