2. 淄博市中心医院口腔科
舌癌是口腔颌面部常见的恶性肿瘤之一, 近年来其发病率有所上升。虽然现阶段以手术为主的综合治疗取得了一定的效果, 但是舌癌易转移, 手术切除难度大, 术后易复发, 手术常常造成病人语言、进食、呼吸等功能的障碍和颜面的畸形[1-2]。目前舌癌的发病机制尚未明确。因此, 在分子层面上揭示舌癌的发病机制, 筛选舌癌发生发展的关键基因, 可能为舌癌的防治提供重要的靶点和标志物。基因芯片是一种高通量获取生物信息的技术, 能高效检测并分析肿瘤组织和正常组织差异表达基因(DEGs)[3]。本研究拟通过分析基因表达数据库(GEO)提供的舌鳞状细胞癌(简称鳞癌)相关基因芯片数据, 筛选DEGs, 并对DEGs进行功能富集分析, 构建蛋白相互作用网络, 同时对关键基因进行生存分析, 为进一步在分子水平研究舌鳞癌的发生发展机制, 为舌鳞癌的诊断和治疗提供一定的理论依据。现将结果报告如下。
1 资料和方法 1.1 数据检索在GEO(https://www.ncbi.nlm.nih.gov/geo)中检索“tongue cancer”, 下载舌鳞癌基因芯片数据。每个数据集均需符合以下条件:①实验用人舌鳞癌组织和正常组织进行比较; ②数据集来自全基因组RNA表达芯片。
1.2 获得DEGs应用R语言(https://www.r-project.org/)对基因芯片原始数据进行注释和过滤, 用Bioconductor( http://bioconductor.org/)提供的RMA算法对各原始芯片数据进行背景校正及归一化等预处理。采用Limma程序包对肿瘤组织和正常组织样本的基因表达值进行比对, 以表达倍数变化值的对数值(log2FC)绝对值>1且调整后P < 0.05为阈值, 获得各数据集的DEGs。采用韦恩图(http://bioinformatics.psb.ugent.be/webtools/Venn/)的方法获取各数据集的共同DEGs。
1.3 GO和KEGG富集分析利用DAVID 6.8数据库(https://david.ncifcrf.gov/)对各数据集的共同DEGs进行GO分析和KEGG分析。
1.4 蛋白相互作用网络分析利用STRING 11.0数据库(https://string-db.org/)对各数据集共同DEGs编码蛋白的相互作用进行网络分析。应用Cytoscape_v3.6.1软件插件Cytohubba寻找蛋白相互作用网络中与舌鳞癌发生发展相关的关键基因。
1.5 关键基因生存分析利用KM plotter网上分析工具(http://kmplot.com/analysis/)分析DEGs表达与头颈部鳞癌病人生存期的相关性, 评价通过生物信息学方法找到的关键基因对疾病预后的预测能力。登录KM plotter网站, 输入基因名称, 选择疾病类别, 样本数设为499, cut off值设为median, 绘制生存曲线, 筛选有显著统计学意义的结果。统计学处理的结果以P < 0.05表示差异有统计学意义。
2 结果 2.1 数据库检索共检索到3个数据集(GSE31056、GSE78060、GSE34105), 其中GSE31056数据集包含24个正常样本和24个肿瘤样本; GSE78060数据集包含4个正常样本和26个肿瘤样本; GSE34105数据集包含16个正常样本和62个肿瘤样本。
2.2 DEGs分析GSE31056、GSE78060和GSE34105基因芯片数据集分别筛选出DEGs为2 193、4 727和3 099个(图 1A~C)。为了减少DEGs筛选结果的假阳性率, 采用韦恩图取交集的方法确定3个数据集的共同DEGs为297个(图 1D)。
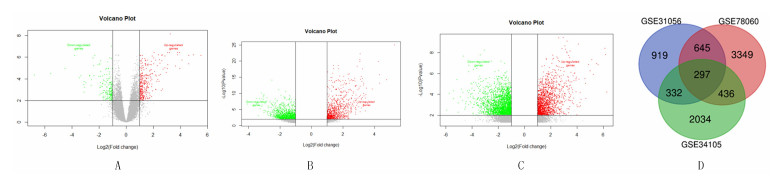 |
| A~C:分别为GSE31056、GSE78060、GSE34105数据集火山图DEGs表达情况, 左侧绿点为低表达基因, 右侧红点为高表达基因; D:3个数据集用韦恩图法取共同DEGs。 图 1 数据库中DEGs表达分析 |
GO分析包括生物学过程(BP)、细胞组分(CC)和分子功能(MF)3部分。DEGs的BP主要富集于血管新生、细胞黏附、氧化还原过程、正向调控转录RNA聚合酶Ⅱ启动子; CC主要富集于朊蛋白细胞外基质、细胞外间隙、细胞外泌体、细胞外组分、细胞表面; MF主要富集于金属内肽酶活化、RNA聚合酶Ⅱ转录因子活性、序列特异性DNA结合、血红蛋白结合、蛋白结合。KEGG分析显示, 通路主要富集于细胞外基质受体相互作用通路、黏着斑通路、小细胞肺癌通路、Toll样受体信号通路和代谢通路。见表 1。
| 表 1 共同DEGs的GO分析和KEGG分析 |
|
|
设置最低要求的相互作用分数为0.4, 得到297个共同DEGs编码蛋白的相互作用关系图(图 2)。采用Cytoscape_v3.6.1软件的插件MCODE对该蛋白相互作用网络进行分析, 得到了由34个共同DEGs构成的核心模块(图 3)。对该核心模块进行GO和KEGG分析, 发现其参与的重要BP主要是细胞黏附、正向调控基因表达作用, 参与的主要信号通路有代谢和PI3K-Akt信号通路等。利用Cytoscape_v3.6.1软件插件cytohubba, 采用Degree算法得到25个在共同DEGs编码蛋白相互作用网络中的关键节点基因(图 4)。
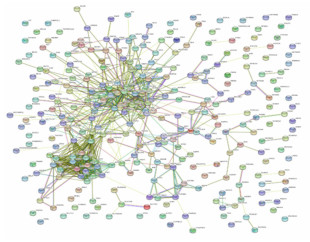 |
| 图 2 共同DEGs编码蛋白相互作用网络图 |
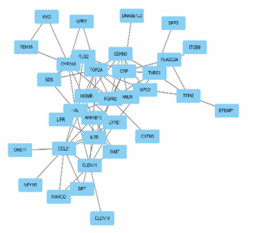 |
| 图 3 MCODE插件确认的网络核心模块图 |
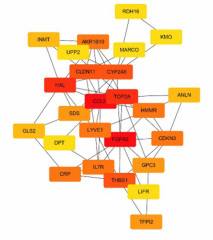 |
| 图 4 cytohubba插件确认的关键节点图 |
利用KM plotter网上分析工具分析关键基因表达与头颈部鳞癌病人生存期的相关性, 结果显示, THBS1(HR=1.19~2.42, P < 0.01)、CRP(HR=1.70~2.03, P < 0.01)、HMMR(HR=1.10~1.88, P < 0.01)、TFPI2(HR=1.00~1.73, P < 0.05)、SDS(HR=1.03~1.80, P < 0.05)、ANLN(HR=1.03~1.79, P < 0.05)基因的表达与头颈部鳞癌病人的预后显著相关, 这6个关键基因表达越高, 病人预后越差(图 5)。
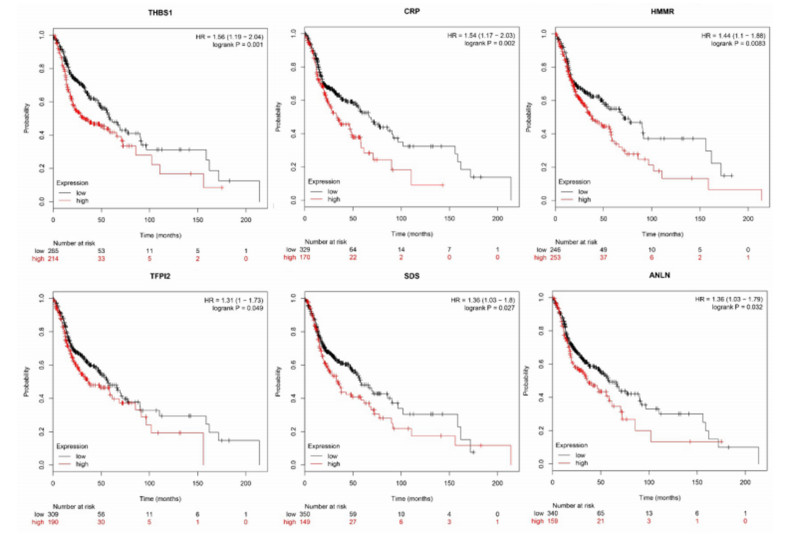 |
| 图 5 关键基因表达与头颈部鳞癌病人生存分析图 |
本研究对GEO中检索到的3个舌鳞癌相关基因芯片数据集进行DEGs筛选, 并对DEGs功能进行富集分析, 构建蛋白相互作用网络分析关键基因表达与头颈部鳞癌病人生存期的相关性, 最终筛选出6个与舌鳞癌发生发展密切相关的关键基因。
THBS1是血小板反应蛋白家族中第一个被识别的蛋白, 在肿瘤微环境中起重要作用。THBS1最开始是作为细胞黏附蛋白而被人们所关注, 很多研究发现THBS1可以在不同种群的多种细胞中调节细胞黏附[4]。最近的研究发现, THBS1表达上调可以增强肿瘤侵袭及转移, 在乳癌中, THBS1FAK信号通路与Hippo信号通路交互作用来调控YAP相关的肿瘤侵袭。鉴于其在肿瘤进展中的重要作用, THBS1有望成为肿瘤治疗的靶点[5]。
CRP即C反应蛋白, 是肝脏产生的血液检查炎性标志物, 在炎症状态时表达升高, 在宿主防御感染过程中起到关键作用[6]。PETRZYK等[7]研究表明, 在结直肠癌中, 肥胖相关的慢性炎症可以通过活化JAK/STAT、MAPK、PI3K、mTOR等信号通路来诱导肿瘤细胞增殖、侵袭和转移。还有研究结果表明, 炎症状态和血浆CRP升高对多种肿瘤产生影响, 炎性标志物表达与结直肠癌病人转移生存率呈负相关[8]。
HMMR是细胞外基质的主要成分, 可以调节细胞运动和细胞周期[9]。STEVENS等[10]报道, 在原发性肺腺癌中HMMR呈高表达。WANG等[11]报道, 在乳癌中RHAMM呈过表达, RHAMM高表达与较差预后评估值相一致。HMMR参与了调节肿瘤细胞生长、侵袭和转移, 其高表达提示肿瘤病理分期更差、预后更差。
TFPI2的生物学功能尚没有完全确定。有学者认为TFPI2作为视网膜色素上皮细胞和血管平滑肌细胞的有丝分裂原来发挥作用[12-13]。在癌症生物学背景下, 关于TFPI2的研究主要集中在其蛋白酶抑制剂活性上。TFPI2结构中包含3个串联重复Kunitz-type蛋白酶抑制域[14]。由于能广泛抑制丝氨酸蛋白酶(包括纤溶酶、胰蛋白酶、糜蛋白酶), TFPI2可以在调节细胞外基质重塑中发挥关键作用[14]。通过广泛抑制蛋白酶, TFPI2可以保护细胞外基质免受降解, 从而抵抗肿瘤侵袭和转移。相关研究表明, TFPI2过表达可以抑制肺癌、前列腺癌、胰腺癌等多种肿瘤细胞生长和侵袭。TFPI2还被发现可以诱导凋亡, 抑制血管新生。因此, TFPI2被认为是一个抑癌因子。后来有研究发现, TFPI2在鳞癌细胞及组织中专一性过表达, 与其他类型的卵巢癌相比, 卵巢鳞癌在Ⅰ期的诊治率很高[15]。本研究舌鳞癌组织中的TFPI2也同样是表达上调, 对病人的预后产生影响。
SDS具有将丝氨酸转化为丙酮酸的作用, 广泛分布于细菌、真菌、动植物的器官中[16]。参与氨基酸代谢的酶一般有两种或多种异构体, 而其中一种异构体会在肿瘤细胞中表达。虽然cSDS基因在肿瘤细胞中转录表达, 很少翻译为蛋白, 但是其在肿瘤细胞中的表达仍有很重要的生理意义[17]。
ANLN是编码蛋白基因, 在大脑、胎盘、睾丸中的表达水平较高, 在心脏、肾脏、肝脏、肺脏、胰腺、前列腺、脾脏中的表达水平较低[18]。研究结果表明, ANLN在多种肿瘤中呈高表达, 如乳癌、结直肠癌、肝癌、胰腺癌等[19]。ANLN高表达是结直肠癌、胃癌、乳癌、头颈癌预后不良的因素[20]。
本研究采用生物信息学的方法分析了舌鳞癌DEGs的相关数据, 筛选了其相关的MF和信号通路, 使我们更深入地了解了舌鳞癌潜在分子发生发展机制, 并为进一步的实验研究提供了理论依据。
| [1] |
SPIOTTO M T, JEFFERSON G, WENIG B, et al. Diffe- rences in survival with surgery and postoperative radiotherapy compared with definitive chemoradiotherapy for oral cavity cancer a national cancer database analysis[J]. JAMA Otola- ryngology Head & Neck Surgery, 2017, 143(7): 691-699. |
| [2] |
CHANG J H, WU C C, YUAN K S, et al. Locoregionally recurrent head and neck squamous cell carcinoma:incidence, survival, prognostic factors, and treatment outcomes[J]. Oncotarget, 2017, 8(33): 55600-55612. |
| [3] |
YOU Lukuan, ZHENG Xuan, HU Yi. Bioinformatics analysis of potential crucial genes in lung squamous cell cancer[J]. Acad J Chin PLA Med Sch, 2018, 39(10): 903-909. |
| [4] |
HUANG Tingting, SUN Li, YUAN Xianglin, et al. Thrombospondin-1 is a multifaceted player in tumor progression[J]. Oncotarget, 2017, 8(48): 84546-84558. |
| [5] |
SHEN Jie, CAO Beibei, WANG Yatao, et al. Hippo component YAP promotes focal adhesion and tumour aggressiveness via transcriptionally activating THBS1/FAK signalling in breast cancer[J]. Journal of Experimental & Clinical Cancer Research, 2018, 37: 175. |
| [6] |
BLACK S, KUSHNER I, SAMOLS D. C-reactive protein[J]. J Biol Chem, 2004, 279(47): 48487-48490. DOI:10.1074/jbc.R400025200 |
| [7] |
PIETRZYK L, TORRES A, MACIEJEWSKI R. Obese-rela- ted chronic low-grade inflammation in promotion of colorectal cancer development[J]. Asian Pac J Cancer, 2015, 16(10): 4161-4168. DOI:10.7314/APJCP.2015.16.10.4161 |
| [8] |
HEIKKIL K, EBRAHIM S, LAWLOR D A. A systematic review of the association between circulating concentrations of C reactive protein and cancer[J]. Journal of Epidemiology and Community Health, 2007, 61(9): 824-833. DOI:10.1136/jech.2006.051292 |
| [9] |
SOHR S, ENGELAND K. RHAMM is differentially expressed in the cell cycle and down-regulated by the tumor suppressor p53[J]. Cell Cycle, 2008, 7(21): 3448-3460. DOI:10.4161/cc.7.21.7014 |
| [10] |
STEVENS L E, CHEUNG W K, ADUA S J, et al. Extracellular matrix receptor expression in subtypes of lung adenocarcinoma potentiates outgrowth of micrometastases[J]. Cancer Research, 2017, 77(8): 1905-1917. DOI:10.1158/0008-5472.CAN-16-1978 |
| [11] |
WANG C, THOR A D, MOORE D H. The overexpression of RHAMM, a hyaluronan-binding protein that regulates ras signaling, correlates with overexpression of mitogen-activated protein kinase and is a significant parameterin breast cancer progression[J]. Clinical Cancer Research, 1998, 4(3): 567-576. |
| [12] |
SHINODA E, YUI Y, HATTORI R, et al. Tissue factor pathway inhibitor-2 is a novel mitogen for vascular smooth muscle cells[J]. Journal of Biological Chemistry, 1999, 274(9): 5379-5384. DOI:10.1074/jbc.274.9.5379 |
| [13] |
TANAKA Y, UTSUMI J, MATSUI M. Purification, mole- cular cloning, and expression of a novel growth-promoting factor for retinal pigment epithelial cells, REF-1/TFPI-2[J]. Invest Ophthalmol Visual Sci, 2004, 45(1): 245-252. DOI:10.1167/iovs.03-0230 |
| [14] |
BAJAJ S, BIRKTOFT J, STEER A. Structure and biology of tissue factor pathway inhibitor[J]. Thromb Haemost, 2001, 86(4): 959-972. |
| [15] |
ARAKAWA N, MIYAGI E, NOMURA A. Secretome-based identification of TFPI2, a novel serum biomarker for detection of ovarian clear cell adenocarcinoma[J]. Journal of Proteome Research, 2013, 12(10): 4340-4350. DOI:10.1021/pr400282j |
| [16] |
HIROFUMI O, TOMOHARU G, MIKIO N. Structure and function relationships of serine dehydratases from various sources[J]. Trends Comp Biochem Physiol, 2000, 6: 1-19. |
| [17] |
HIROFUMI O, TOMOHARU G, MIKIO N. Enzymatic and biochemical properties of a novel human serine dehydratase isoform[J]. Biochimica Et Biophysica Acta, 2006, 1764(5): 961-967. DOI:10.1016/j.bbapap.2006.02.010 |
| [18] |
OEGEMA K, SAVOIAN M S, MITCHISON T J. Functional analysis of a human homologue of the drosophila actin binding protein anillin suggests a role in cytokinesis[J]. Journal of Cell Biology, 2000, 150(3): 539-552. DOI:10.1083/jcb.150.3.539 |
| [19] |
ZHOU Weibing, WANG Zhan, SHEN Ni, et al. Knockdown of ANLN by lentivirus inhibits cell growth and migration in human breast cancer[J]. Molecular and Cellular Biochemistry, 2015, 398(1/2): 11-19. |
| [20] |
XIA Leilei, SU Xiaoling, SHEN Jizi. ANLN functions as a key candidate gene in cervical cancer as determined by integra- ted bioinformatic analysis[J]. Cancer Management and Research, 2018, 10: 663-670. DOI:10.2147/CMAR.S162813 |
 2019, Vol. 55
2019, Vol. 55

