肾细胞癌(RCC)是最常见的肾脏癌症,占所有成人恶性肿瘤的2%~3%[1-2]。有20%~30%的病人被诊断为RCC时已经发现远处转移。此外,另有20%的病人接受肾切除术后会复发并发展成为转移性RCC [3-4]。转移性RCC病人生存率低,预后极差[5-6]。乳头状肾细胞癌(PRCC)是第二常见的RCC类型,占所有RCC的10%~20%[1]。PRCC可根据组织病理学特征进一步分为两种主要亚型[7-9]。癌症基因组图谱(TCGA)是临床信息和基因测序数据的大型综合集合。既往RCC研究主要以透明细胞RCC为主,缺乏PRCC相关研究。为了探究PRCC涉及的信号传导途径和驱动基因并且进行系统分析,本文利用TCGA进行数据挖掘,筛选PRCC中差异表达基因及富集的信号通路。现将结果报告如下。
1 材料与方法 1.1 实验样本信息收集于TCGA数据库(https://cancergenome.nih.gov/)中收集与PRCC相关的RNA的测序数据(Level3),包括289例PRCC组织和32例癌旁非癌肾组织。
1.2 筛选PRCC中异常表达的mRNAs利用genecode数据库筛选mRNA测序数据,共涵盖20 271个mRNA数据。随后以R语言包edgeR计算异常表达的mRNAs(差异倍数>2并且错误发现率(FDR)校正后P值< 0.01)。排除超过75%的样品中表达<2的mRNA。
1.3 基因本体分析和富集途径分析使用在线数据库DAVID(https://david.ncifcrf.gov/)进行差异基因的基因本体分析和富集途径分析。基因本体分析和富集途径分析均以P值< 0.05作为临界标准。
1.4 基因集富集分析基因组富集分析使用Java GSEA软件。基因组富集分析软件及注释基因集均由Broad研究所网站(http://www.broadinstitute.org/gsea/index.jsp)下载。基因集P < 0.05并且FDR < 0.25被认为具有统计学差异。
1.5 统计学处理使用R语言(版本号3.5.1)中的edgeR包筛选PRCC和癌旁正常组织中不同表达的基因,筛选标准为FDR校正后P值< 0.01和差异倍数>2的基因。对于GO和KEGG途径分析,其中P < 0.05的GO术语或途径是有统计学差异的。
2 结果 2.1 癌与癌旁配对样本差异表达的mRNA对PRCC标本及癌旁正常组织的mRNA测序数据进行差异基因分析(差异倍数>2并且FDR校正后P值< 0.01),筛选4 639个差异表达的基因,在样品中2 638个mRNA转录物被上调并且2 001个mRNA转录物被下调。火山图见图 1。其中,PRCC及其癌旁正常组织中50种最具有表达差异的mRNA(从倍数变化最大到最小列出)见表 1。
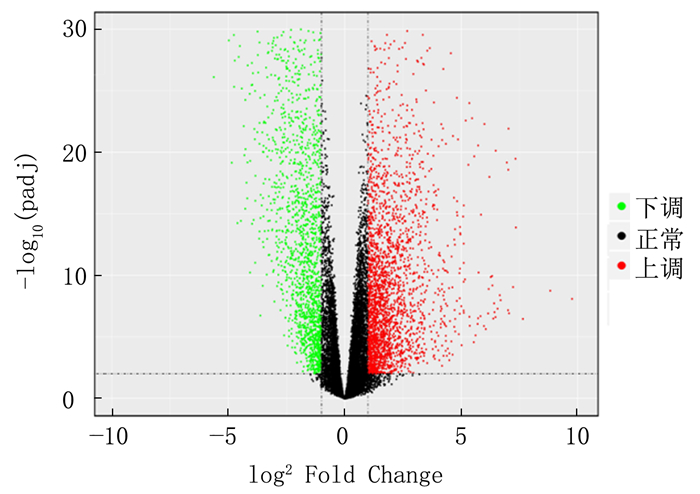 |
| 图 1 PRCC及其癌旁正常组织的mRNA表达热图 |
| 表 1 PRCC及其癌旁正常组织50种最具有表达差异的mRNA |
|
|
利用在线基因功能分类工具(https://david.ncifcrf.gov/gene2gene.jsp),基于功能相似性对筛选出的50种最具有表达差异的基因进行分组。上调组筛选出1个功能相关基因组,基因为SIGLEC8、CNTN6、VSTM2A、VSTM2L。下调组筛选出2个功能相关基因组,其中一个基因组为SLC34A3、SCNN1B、SLC22A8、SLC9A4、SLC12A1,另外一个基因组为AQP2、MUC15、SLC22A8、SLC14A2。检索相关文献,我们发现SIGLEC8表达的增强预测了PRCC病人的不良预后[10]。另外,MUC15在PRCC中的表达降低[11],但是其余大部分基因在PRCC中的作用仍不明确。
2.2 差异表达基因的基因本体分析利用在线数据库DAVID分析差异表达基因,以P < 0.05作为临界标准。基因本体分析分为3个功能组:分子功能组、生物过程组和细胞成分组。在生物过程组中,上调的基因主要富集于信号转导、免疫反应、炎症反应等,下调基因主要富集于信号转导、细胞黏附等。在细胞成分组中,上调基因主要富集在膜的组成部分、质膜、细胞外区域等,下调基因主要富集于膜的组成部分、质膜等。在分子功能组中,上调基因主要富集于钙离子结合、受体结合等,下调基因主要富集于钙离子结合、蛋白质同源二聚化活性等。其中,PRCC标本转录组上调基因最显著富集的5个基因本体术语见图 2A~C,下调基因最显著富集的5个基因本体术语见图 2D~F。
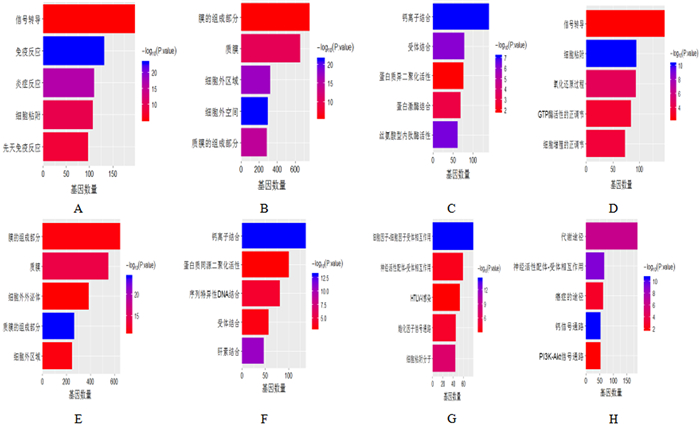 |
| A、D:转录组上调、下调基因最显著富集的5个生物学过程的基因本体术语;B、E:转录组上调、下调基因最显著富集的5个细胞成分的基因本体术语;C、F:转录组上调、下调基因最显著富集的5个分子功能的基因本体术语。G:上调基因最显著富集的5个信号通路;H:下调基因最显著富集的5个信号通路。 图 2 差异表达基因的基因本体分析及信号通路富集分析 |
使用KEGG PATHWAY在线网站进行差异基因的功能和信号传导途径富集。上调的基因主要富集于细胞因子-细胞因子受体相互作用、神经活性配体-受体相互作用等,下调的基因主要富集于代谢途径、神经活性配体-受体相互作用、癌症的途径等。见图 2G、H。
2.4 基因集富集分析基因集富集分析通过关注具有共同生物功能、染色体定位或调节的基因组,从而筛选出可能与疾病表型相关的过量表达的基因或蛋白质。我们使用KEGG基因组作为基因集对PRCC及其癌旁正常组织的转录组数据进行基因集富集分析。确定了7个上调途径以及12个下调途径。见图 3A、B。
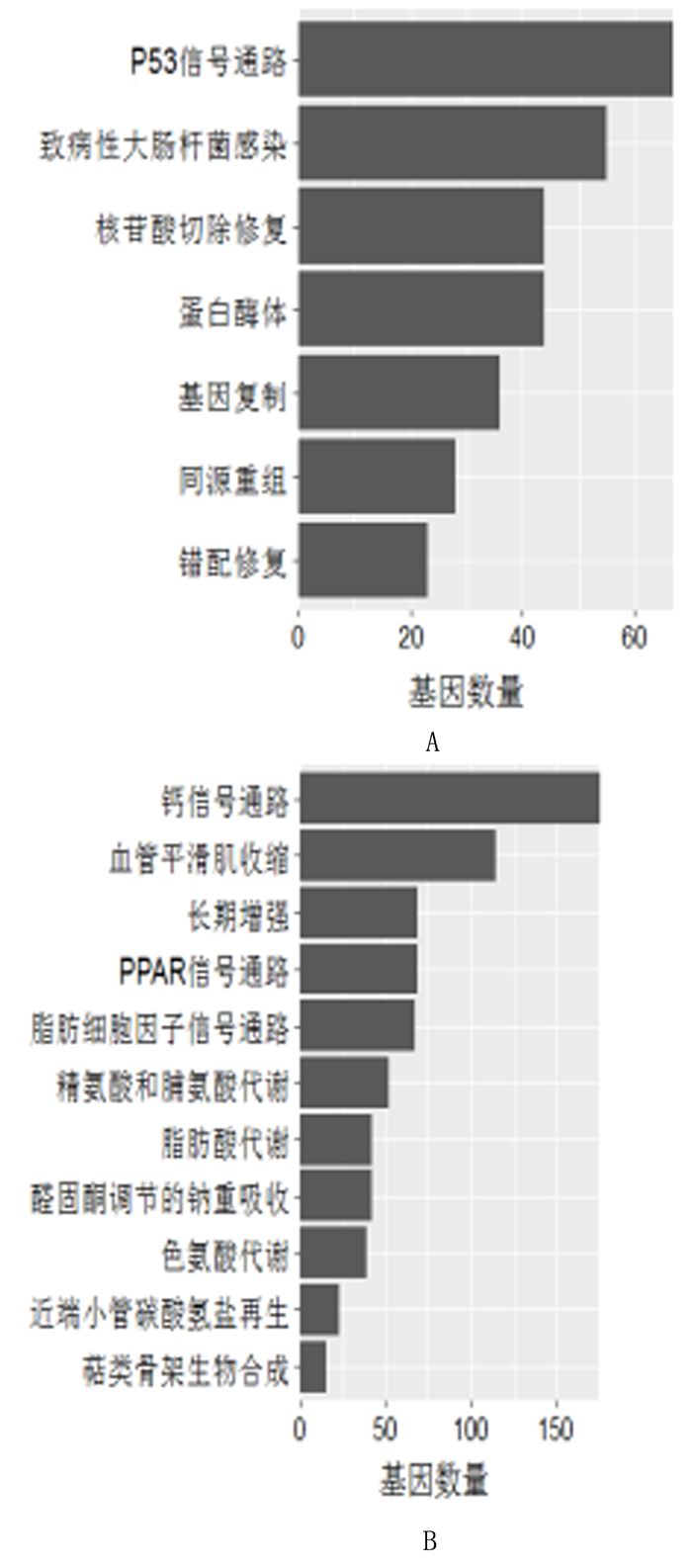 |
| A:上调组基因集富集分析;B:下调组基因集富集分析。 图 3 基因集富集分析 |
在过去的几十年中,已经进行了大量的基础和临床研究,揭示PRCC形成和发展的原因和潜在机制[12-15]。已报道PRCC具有代表性遗传基因突变和失调的途径包括:MET基因突变、HIPPO途径、NRF2途径、染色质修饰基因突变等[9]。本研究应用生物信息分析技术深入分析TCGA数据集,并在第一步中确定了4 639个差异表达的基因(2 638个上调和2 001个下调)。在第二步中,对筛选差异表达基因进行基因本体分析及代谢通路分析,本研究中筛选的上调基因主要富集在信号转导、免疫反应、炎症反应、膜的组成部分和钙离子结合等途径中;下调基因主要富集在信号转导、细胞黏附、膜的组成部分和钙离子结合等途径中。改变的基因和信号转导途径为理解PRCC的分子基础提供相关数据,从细胞通路及代谢途径更好揭示PRCC内在分子机制,探究预后的生物标志物及治疗靶点。既往研究已提示上皮细胞黏附及其活化分子在高级别RCC中的过度表达可能是预后的有用标志[16]。在PRCC发生骨转移病人的组织样本和原代细胞中,检测到钙离子受体表达明显增强[17-18]。本研究中,同样观察到钙离子结合途径发生明显改变,与有关文献报道相符合。以前研究表明,肿瘤的发生与炎症状态有密切关系[19],这与本研究显示免疫反应、炎症反应相关基因集在PRCC上调具有一致性。此外,既往研究发现肿瘤可建立肿瘤内免疫抑制环境,以支持其生长并促进免疫逃避[20-21],本研究结果同样显示肿瘤内免疫反应的改变。肿瘤内的代谢物水平的增加可以促进癌症的发生和发展,可导致细胞信号、酶活性或代谢通量的改变[22]。散发性和家族性2型PRCC检测到增强的有氧糖酵解,并且具有抗氧化反应的细胞表型[23-24]。
本研究第三步中,利用基因集富集分析方法分析筛选的差异表达基因的代谢途径,结果表明P53信号通路和DNA修复途径在PRCC中上调。DNA损伤的修复在维持基因组完整性方面起着关键作用[25]。多种常规DNA修复途径的多功能DNA修复中心经常在癌症中被改变[26]。有研究表明,DNA损伤修复途径中不利等位基因数量的增加,会增加RCC风险[27]。不过也有证据表明,DNA修复基因多态性可能不会影响RCC的易感性,但可能会导致DNA修复能力的改变,从而影响肿瘤的进展[28]。肿瘤抑制基因p53是人类癌症中最常见的突变基因,在细胞凋亡、基因组稳定性和抗血管生成中发挥作用,可以介导RCC细胞周期停滞和细胞凋亡[29]。研究表明,P53阳性表达与RCC病人预后不良和晚期临床病理特征相关,这表明P53是一个潜在有效的治疗靶点[30]。
综上所述,利用生物信息学分析,我们共筛选了4 639个候选基因,其富集于信号转导、钙离子结合、细胞因子-细胞因子受体相互作用、代谢途径、P53信号通路和DNA修复等途径。这些发现可提高我们对PRCC病因和潜在分子事件的理解,这些候选基因和途径有可能成为PRCC的治疗靶点。
| [1] |
COURTHOD G, TUCCI M, MAIO M D, et al. Papillary renal cell carcinoma:a review of the current therapeutic landscape[J]. Crit Rev Oncol Hematol, 2015, 96(1): 100-112. DOI:10.1016/j.critrevonc.2015.05.008 |
| [2] |
SIEGEL R L, MILLER K D. Cancer statistics, 2019[J]. CA-Cancer J Clin, 2019, 96(1): 7-34. |
| [3] |
LJUNGBERG B, CAMPBELL S C, CHO H Y, et al. The epidemiology of renal cell carcinoma[J]. European Urology, 2011, 60(4): 615-621. DOI:10.1016/j.eururo.2011.06.049 |
| [4] |
KING S C, POLLACK L A, LI J, et al. Continued increase in incidence of renal cell carcinoma, especially in young patients and high grade disease:United States 2001 to 2010[J]. J Urol, 2014, 191(6): 1665-1670. DOI:10.1016/j.juro.2013.12.046 |
| [5] |
SHUCH B, HAHN A W, AGARWAL N. Current treatment landscape of advanced papillary renal cancer[J]. J Clin Oncol, 2017, 35(26): 2981-2983. DOI:10.1200/JCO.2017.74.3328 |
| [6] |
CONNOR W J, DONSkOV F, FRACCON A P, et al. Cha-racterizing the outcomes of metastatic papillary renal cell carcinoma[J]. Cancer Medicine, 2017, 6(5): 902-909. DOI:10.1002/cam4.1048 |
| [7] |
ALGABA F, AKAZA H, LOPEZ-BELTRAN A, et al. Current pathology keys of renal cell carcinoma[J]. Eur Urol, 2011, 60(4): 634-643. DOI:10.1016/j.eururo.2011.06.047 |
| [8] |
KLATTE T, PANTUCK A J, SAID J W, et al. Cytogenetic and molecular tumor profiling for type 1 and type 2 papillary renal cell carcinoma[J]. Clin Cancer Res, 2009, 15(4): 1162-1169. DOI:10.1158/1078-0432.CCR-08-1229 |
| [9] |
LINEHAN W M, SPELLMAN P T, RICKETTS C J, et al. Comprehensive molecular characterization of papillary renal-cell carcinoma[J]. N Engl J Med, 2016, 374(2): 135-145. DOI:10.1056/NEJMoa1505917 |
| [10] |
OU C, LIU L, WANG J, et al. Enhancement of Siglec-8 expression predicts adverse prognosis in patients with clear cell renal cell carcinoma[J]. Urol Oncol, 2017, 35(10): 607-607. |
| [11] |
KING R J, YU F, SINGH P K. Genomic alterations in mucins across cancers[J]. Oncotarget, 2017, 8(40): 67152-67168. |
| [12] |
ALBIGES L, GUEGAN J, FORMAL A L, et al. MET is a potential target across all papillary renal cell carcinomas:result from a large molecular study of pRCC with CGH array and matching gene expression array[J]. Clin Cancer Res, 2014, 20(13): 3411-3421. DOI:10.1158/1078-0432.CCR-13-2173 |
| [13] |
LIU K, REN Y, PANG L, et al. Papillary renal cell carcinoma:a clinicopathological and whole-genome exon sequencing study[J]. Int J Clin Exp Pathol, 2015, 8(7): 8311-8335. |
| [14] |
KOVAC M, NAVAS C, HORSWELL S, et al. Recurrent chromosomal gains and heterogeneous driver mutations cha-racterise papillary renal cancer evolution[J]. Nat Commun, 2015, 6(1): 6336. |
| [15] |
DURINCK S, STAWISKI E W, PAVIA-JIMENEZ A, et al. Spectrum of diverse genomic alterations define non-clear cell renal carcinoma subtypes[J]. Nat Genet, 2015, 47(1): 13-21. DOI:10.1038/ng.3146 |
| [16] |
ZIMPFER A, MARUSCHKE M, REHN S, et al. Prognostic and diagnostic implications of epithelial cell adhesion/activating molecule(EpCAM)expression in renal tumours:a retrospective clinicopathological study of 948 cases using tissue microarrays[J]. BJU Int, 2014, 114(2): 296-302. |
| [17] |
BRENNER W, HABER T, JUNKER K, et al. Bone metastasis by renal cell carcinoma. Importance of Calcium and calcium-sensing receptor[J]. Urologe A, 2015, 54(6): 839-843. DOI:10.1007/s00120-014-3716-9 |
| [18] |
FREES S, BREUKSCH I, HABER T, et al. Calcium-sensing receptor(CaSR)promotes development of bone metastasis in renal cell carcinoma[J]. Oncotarget, 2018, 9(21): 15766-15779. |
| [19] |
KORNILUK A, KOPER O, KEMONA H, et al. From inflammation to cancer[J]. Ir J Med Sci, 2017, 186(1): 57-62. DOI:10.1007/s11845-016-1464-0 |
| [20] |
BUSSE A, ASEMISSEN A, NONNENMACHER A, et al. Systemic immune tuning in renal cell carcinoma:favorable prognostic impact of TGF-beta1 mRNA expression in perip-heral blood mononuclear cells[J]. J Immunother, 2011, 34(1): 113-119. |
| [21] |
BINNEWIES M, ROBERTS E W, KERSTEN K, et al. Understanding the tumor immune microenvironment (TIME) for effective therapy[J]. Nature Medicine, 2018, 24(5): 541-550. DOI:10.1038/s41591-018-0014-x |
| [22] |
SULLIVAN L B, GUI D Y, VANDERHEIDEN M G. Altered metabolite levels in cancer:implications for tumour biology and cancer therapy[J]. Nat Rev Cancer, 2016, 16(11): 680-693. DOI:10.1038/nrc.2016.85 |
| [23] |
OOI A, WONG J C, PETILLO D, et al. An antioxidant response phenotype shared between hereditary and sporadic type 2 papillary renal cell carcinoma[J]. Cancer Cell, Oct 18, 2011, 20(4): 511-523. DOI:10.1016/j.ccr.2011.08.024 |
| [24] |
SRINIVASAN R, RICKETTS C J, SOURBIER C, et al. New strategies in renal cell carcinoma:targeting the genetic and metabolic basis of disease[J]. Clin Cancer Res, 2015, 21(1): 10-17. DOI:10.1158/1078-0432.CCR-13-2993 |
| [25] |
MARGULIS V, LIN J, YANG H, et al. Genetic susceptibility to renal cell carcinoma:the role of DNA double-strand break repair pathway[J]. Cancer Epidemiol Biomarkers Prev, 2008, 17(9): 2366-2373. DOI:10.1158/1055-9965.EPI-08-0259 |
| [26] |
DIETLEIN F, THELEN L, REINHARDT H C. Cancer-specific defects in DNA repair pathways as targets for personalized therapeutic approaches[J]. Trends Genet, 2014, 30(8): 326-339. DOI:10.1016/j.tig.2014.06.003 |
| [27] |
LIN J, PU X, WANG W, et al. Case-control analysis of nucleotide excision repair pathway and the risk of renal cell carcinoma[J]. Carcinogenesis, 2008, 29(11): 2112-2119. DOI:10.1093/carcin/bgn189 |
| [28] |
SAKANO S, HINODA Y, OKAYAMA N, et al. The association of DNA repair gene polymorphisms with the development and progression of renal cell carcinoma[J]. Ann Oncol, 2007, 18(11): 1817-1827. DOI:10.1093/annonc/mdm337 |
| [29] |
LIU Q J, SHEN H L, LIN J, et al. Synergistic roles of p53 and HIF1alpha in human renal cell carcinoma-cell apoptosis responding to the inhibition of mTOR and MDM2 signaling pathways[J]. Drug Des Devel Ther, 2016, 10(1): 745-755. |
| [30] |
WANG Z, PENG S, JIANG N, et al. Prognostic and clinicopathological value of p53 expression in renal cell carcinoma:a meta-analysis[J]. Oncotarget, 2017, 8(60): 102361-102370. |
 2019, Vol. 55
2019, Vol. 55

