2. 同济大学经济与管理学院;
3. 青岛大学附属医院手术室
肾细胞癌(RCC)是泌尿系统常见肿瘤,在男性肿瘤中居第9位,在女性肿瘤中居第14位。2018年,RCC占全球新发肿瘤病例的2.2%,死亡率为1.8%,其中80%~90%为肾透明细胞癌(ccRCC)[1-2]。尽管癌症的检测和治疗取得了很大进展,但ccRCC的总生存率仍然很低。超过1/3的病人在诊断时已经发生转移,Ⅳ期ccRCC的5年标准化相对生存率仅为6%,而Ⅰ期约为84%[3-4]。因此,研究ccRCC的发生发展机制,寻找新的生物标志物对ccRCC的早期诊断、治疗及预后判断具有重要意义。传统的“单疾病单基因”的研究模式不能从多基因协同角度了解疾病的发生与发展。本研究采用生物信息学的方法,通过加权基因共表达网络分析(WGCNA),将功能相似的基因归入同一基因模块,探究基因之间的相关性以及基因模块和临床特征之间的关联,筛选枢纽基因,以了解ccRCC发生进展过程中的关键基因及信号通路,为ccRCC治疗寻找新的靶点。
1 材料与方法 1.1 差异表达基因的筛选从NCBI的GEO数据库(https://www.ncbi.nlm.nih.gov/geo/)下载编号GSE73731的ccRCC芯片数据集。该数据集基于GLP570平台,包括265例ccRCC组织,其中包括高分化(Furhman Ⅰ/Ⅱ)病例112例,中/低分化病例144例(Furhman Ⅲ/Ⅳ),未分级者9例。Ⅰ期病人41例,Ⅱ期病人12例,Ⅲ期病人28例,Ⅳ期病人44例,分析数据缺失140例。利用R软件中Bioconductor(http://www.bioconductor.org)内的Affy包[5]读取原始文件,使用RMA算法预处理得到标准化的基因表达谱数据。剔除离群样本,用R软件的limma包[6]对基因表达矩阵进行分析,设置校正后P值(FDR)<0.05和对数化表达变化倍数(|log2FC|)>2.0作为筛选表达差异基因的阈值,得到高分化与中/低分化ccRCC组织间的差异表达基因(DEGs),并绘制热图及火山图。
1.2 基因共表达网络构建使用R软件的WGCNA包[7],通过方差分析筛选方差前25%的基因用于WGCNA。计算各基因间的Pearson相关系数,选择适当的软阈值β使得构建的网络更符合无标度网络的标准。采用一步法构建基因网络,将邻接矩阵转化为拓扑重叠矩阵TOM,利用层次聚类产生一个基因的层次聚类树。计算基因显著性(GS)以及模块显著性(MS),用以衡量基因与临床信息的显著性,并分析模块及模型的显著关联。
1.3 枢纽基因的筛选计算各基因模块身份(MM)以衡量基因在模块中的重要性。设置参数为|MM|>0.9和|GS|>0.2筛选基因。将枢纽模块中的基因上传至STRING数据库(https://string-db.org/)构建蛋白相互作用网络,选取点度中心性(degree)>80筛选基因,两者取交集即为枢纽基因。
1.4 枢纽基因的表达与ccRCC分级、分期及预后的关系使用GSE73731数据对枢纽基因进行线性回归分析,探究基因表达量与ccRCC分级之间的关系,并绘制受试者工作特征(ROC)曲线,计算曲线下面积(AUC),当AUC>0.7时,认为该基因对诊断ccRCC进展具有较高的灵敏度和特异度。使用基于TCGA的GEPIA数据库(http://gepia.cancer-pku.cn/)和UALCAN数据库(http://ualcan.path.uab.edu/)分析枢纽基因在ccRCC中的表达水平及其与肿瘤分级、分期、预后的关系。
1.5 GO和KEGG富集分析使用DAVID数据库(http://david.abcc.ncifcrf.gov/)对特定模块的基因进行GO和KEGG通路分析,以错误发现率(FDR)<0.05为差异具有统计学意义。
2 结果 2.1 差异表达基因筛选使用R软件读取及预处理芯片数据后,共得到20 460个表达谱基因。以FDR<0.05和|log2FC|>5.0为阈值筛选后,得到333个DEGs,其中上调基因152个,下调基因181个。DEGs热图及火山图见图 1。
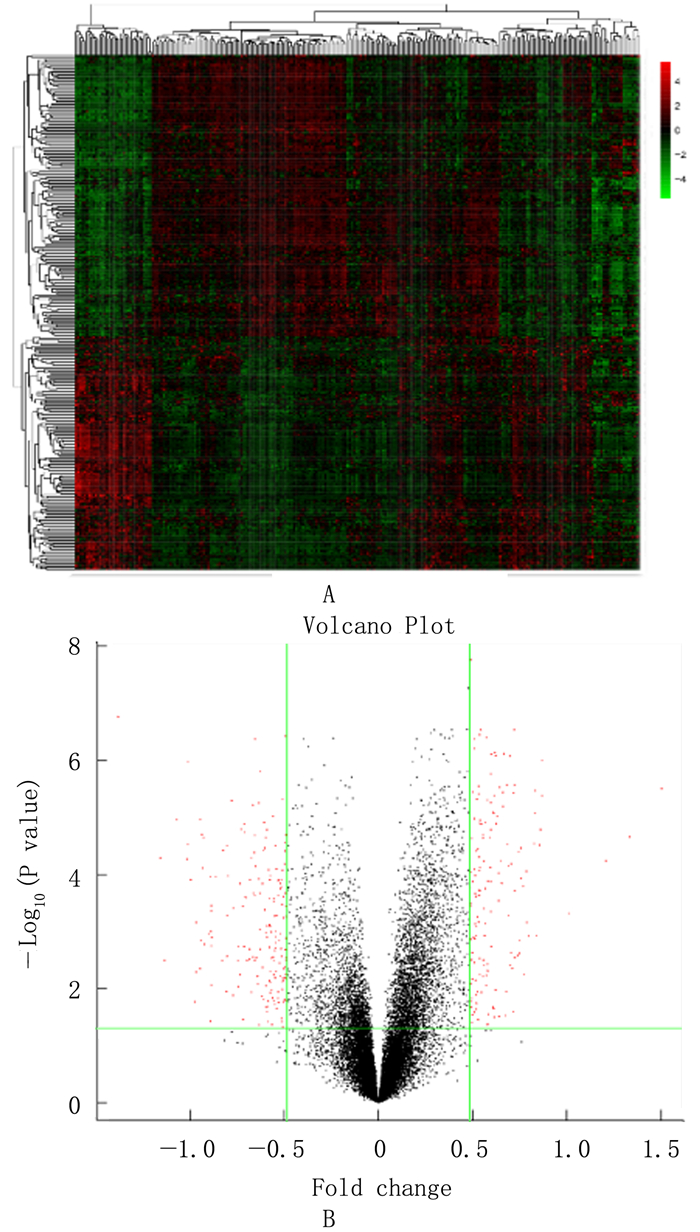 |
| A:火山图;B:热图。 图 1 DEGs热图及火山图 |
本研究通过方差分析筛选方差前25%的基因共5 115个用于WGCNA。以相关系数等于0.9作为标准,使用pickSoftThreshold函数,选择邻接矩阵权重参数(软阈值)β=6构建基因模块。使用一步法构建共表达矩阵,并利用动态混合剪切法,得到12个基因模块, 其中green模块(包括355个基因)的GS高于其他模块(图 2)。因此,green模块与ccRCC病理分级的相关性最高,与肿瘤进展明显相关。
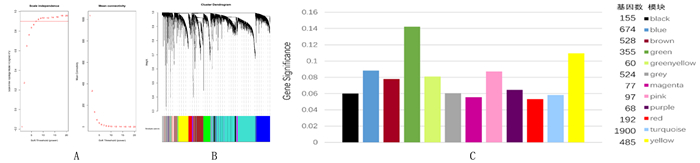 |
| A:软阈值;B:基因模块;C:枢纽模块。 图 2 软阈值β确定、基因模块获取及枢纽模块筛选 |
对green模块基因以|MM|>0.9和|GS|>0.2为参数筛选枢纽基因得到10个枢纽基因,将green模块基因上传至STRING数据库以degree>80为参数筛选枢纽基因得到23个枢纽基因,两者取交集得到CEP55、CCNB1、NUF2、BUB1B、KIF14共5个在共表达网络和蛋白相互作用网络皆重要的枢纽基因(表 1)。
| 表 1 green模块枢纽基因 |
|
|
使用GSE73731数据集进行线性回归分析,结果显示各枢纽基因与ccRCC组织学分级呈正相关(t=17.53~25.18, P < 0.01)。见图 3A~E。ROC曲线分析,BUB1B基因对ccRCC组织学分级具有较高诊断效能(AUC=0.706,P<0.01),而CEP55、CCNB1、NUF2、KIF14对ccRCC组织学分级诊断效能较弱(AUC=0.680~0.691,P<0.01)。见图 3F~J。基于TCGA的UALCAN和GEPIA数据库验证结果显示,5个枢纽基因的表达水平在肿瘤组织中均明显升高(FC=1.76~7.13,P<0.01),且与肿瘤的分级及分期明显相关。见图 4。预后结果显示,CEP55、NUF2、BUB1B与肿瘤的总生存期(OS)明显相关(HR=1.5~1.9,P<0.01)。见图 5A~E。CEP55、CCNB1、BUB1B、KIF14与肿瘤的无病生存期(DFS)明显相关(HR=1.6~1.7,P<0.01)。见图 5F~J。
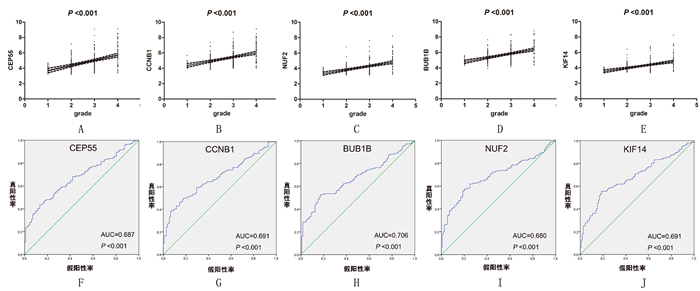 |
| A~E:基因与分级相关性;F~J:基因的诊断效能。 图 3 基于GSE73731数据集的各枢纽基因与ccRCC组织学分级相关性及诊断效能 |
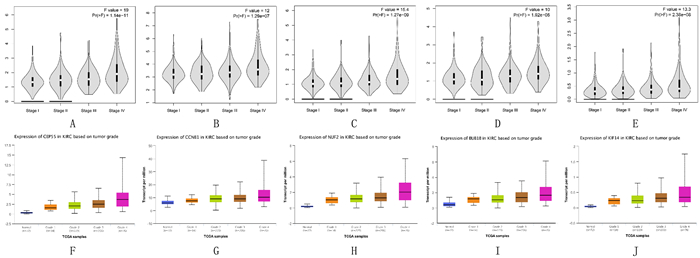 |
| A~E:基因与肿瘤分期关系; F~J:基因与肿瘤分级关系。 图 4 GEPIA和UALCAN数据库中各枢纽基因表达水平与肿瘤分期及分级的关系 |
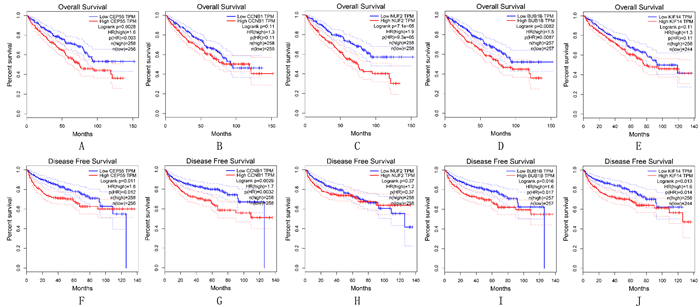 |
| A~E:基因与OS关系; F~J:基因与DFS关系。 图 5 各枢纽基因表达水平与OS及DFS的关系 |
为了了解枢纽模块的可能功能,对green模块中的基因进行了GO和KEGG富集分析。GO富集分析显示,模块基因主要富集在细胞周期、细胞器分裂、核分裂等生物学过程;KEGG富集分析显示,模块基因主要富集在细胞周期通路上。见图 6。
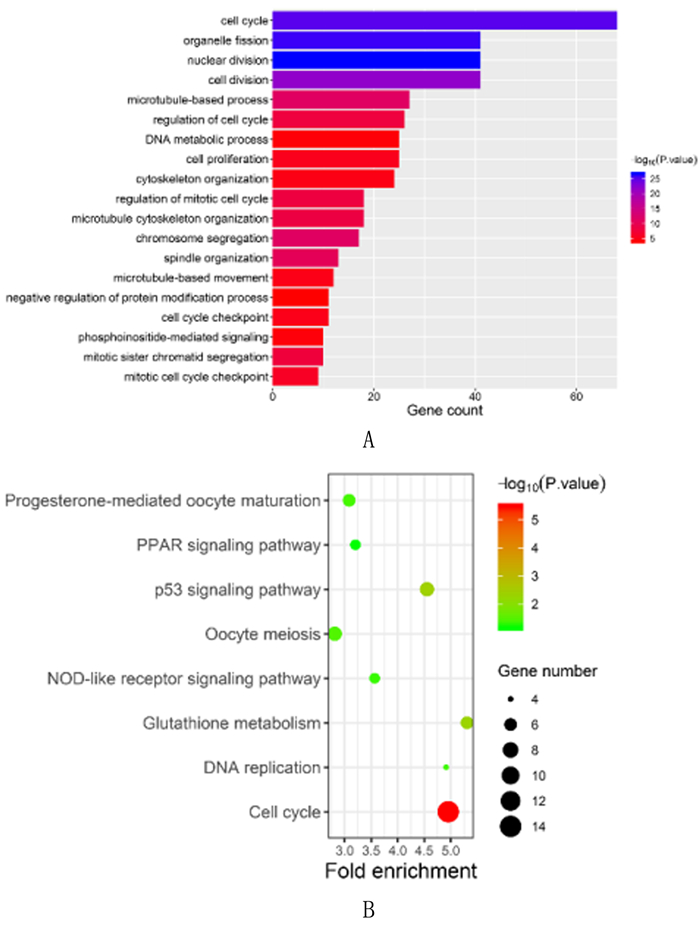 |
| A:GO分析;B:KEGG分析。 图 6 枢纽模块基因的GO和KEGG富集分析 |
本研究利用生物信息学方法,通过对ccRCC芯片数据集GSE73731进行分析,筛选得到表达上调基因152个,表达下调基因181个。通过WGCNA,将表达模式相似的基因进行聚类,并分析模块与特定性状或表型之间关联。结果显示,green模块与ccRCC病理分级的相关性最高,green模块内基因集与肿瘤的进展与预后密切相关。为进一步筛选在ccRCC进展过程中的关键基因,将green模块中进一步筛选出的基因与蛋白相互作用网络筛选出的基因交集,筛选出CEP55、CCNB1、NUF2、BUB1B、KIF14共5个枢纽基因。对GSE73731数据集进行线性回归分析,结果显示各枢纽基因与ccRCC组织学分级密切相关,且BUB1B基因具有较高的诊断价值,说明其能够区分不同病理分级的ccRCC。使用TCGA数据进行独立的外部验证,各枢纽基因的表达水平在肿瘤组织中均明显升高,且与肿瘤的分级及分期明显相关,说明其在ccRCC发生及进展过程中起关键作用。本文预后结果显示,CEP55、BUB1B基因与肿瘤的OS及DFS明显相关,提示CEP55和BUB1B基因对ccRCC病人的预后具有一定的预测价值。
CEP55基因编码一种中心体相关蛋白,在中间体依赖性的细胞功能如中心体复制、细胞周期及胞质分裂的调节中发挥重要作用[8]。有研究表明,在肝细胞肝癌、肺腺癌、卵巢癌、结肠癌、乳癌等肿瘤中,CEP55高表达与肿瘤的高恶性程度、高侵袭性以及不良预后相关[9-10]。CCNB1编码细胞周期蛋白B1,是细胞周期G2/M期的重要调控因子,在非小细胞肺癌、外阴鳞状细胞癌、结直肠癌中对肿瘤病人的抗药性、局部或远处转移、复发、生存等指标具有良好的预测价值[11-13]。细胞分裂相关基因NUF2编码的蛋白,作为NDC80复合体的重要组成部分之一,在动粒-微管黏附中扮演重要角色,在有丝分裂和肿瘤发生发展中起着重要作用[14-16]。BUB1B为纺锤体检测点蛋白,作为有丝分裂检测点的重要功能蛋白,调节细胞周期及有丝分裂。BUB1B在肾癌及乳癌等多种肿瘤中过表达,且其突变及过表达与染色体不稳定性、细胞分化和衰老相关,可促进肿瘤的发生及进展[17-18]。KIF4作为驱动蛋白超家族中的成员,可调节纺锤体的形成、染色体的分离和胞质分裂,其表达异常可引起染色体分离失败和胞质分裂不完全,从而引起细胞异常、增殖和分化,诱发肿瘤形成,其异常表达已经在多种恶性肿瘤中得到证实[19-21]。
本研究对枢纽模块基因进行GO和KEGG富集分析,结果显示,模块基因主要富集在细胞周期等相关生物学过程及通路上。在真核生物中,细胞周期主要受细胞周期蛋白(Cyclin)、细胞周期蛋白依赖激酶(CDK)以及细胞周期蛋白依赖激酶抑制剂(CKI)所构成的Cyclin-CDK-CKI信号网络精准调节。具有明显周期性表达的Cyclin可以与不同的CDK结合成复合物,并激活CDK的激酶活性,从而在不同时相对细胞周期进行调控。而CKI对CDK具有抑制作用。Cyclin-CDK-CKI信号网络对细胞周期的调控主要通过Rb途径和p53途径。在Rb途径中,生长因子与细胞表面受体如fos/jun/myc结合,可促进Cyclin表达并形成Cyclin-CDK复合物,磷酸化Rb蛋白后,Rb-E2F复合物释放E2F进入细胞核,促进下游DNA表达。而CKI可以抑制Cyclin-CDK活性,使得Rb去磷酸化,阻滞细胞周期进展。在p53通路中,DNA受损后,p53可结合到p21基因启动子区,激活p21转录。P21作为重要的CKI,可抑制CDK活性,可阻滞细胞从G1期进入S期[11-12, 22]。
CCNB1是细胞周期中G2/M转换的关键因子,可进入细胞核内,与CDK1结合形成CCNB1-CDK1复合物,通过Rb途径诱导细胞进入M期。当细胞退出M期时,CCNB1降解,CDK1激酶活性丧失,细胞进入下一周期[22]。一项针对骨肉瘤的研究表明,CEP55的表达水平与CCND1呈正相关,敲低CEP55表达导致CCND1表达水平降低。CCND1可与CDK4/CDK6形成复合物,调控G1期到S期的转换[23-24]。一项针对肝细胞肝癌的研究表明,敲低NUF2表达可导致CCNB1、Cdc25A、Cdc2等蛋白表达水平降低,诱导细胞周期停滞在G0/G1期,说明NUF2也可以通过Rb通路调控细胞周期[25]。一项针对多发性骨髓瘤的研究表明,BUB1B可通过介导CDC20/CCNB轴促进细胞增殖,在肿瘤进展中起重要作用[26]。有研究表明,KIF14敲低可下调Skp2和Cks1的表达,进而抑制蛋白酶体依赖性p27Kip1泛素化,p27Kip1的增加抑制细胞周期蛋白的表达,包括CCNB1、CCND1和CCNE1,从而抑制肿瘤发生及进展[25, 27-28]。因此,CEP55、CCNB1、NUF2、BUB1B、KIF14等5个枢纽基因可能通过细胞周期相关通路,尤其是Rb通路,来影响ccRCC的发生、进展及预后。
综上所述,本研究通过构建基因共表达网络,筛选出与ccRCC进展相关的5个枢纽基因,这5个基因与肿瘤的分期、分级及预后密切相关;枢纽基因可能通过细胞周期相关通路来影响ccRCC的发生、进展及预后[1]。
| [1] |
BRAY F, FERLAY J, SOERJOMATARAM I, et al. Global cancer statistics 2018:GLOBOCAN estimates of incidence and mortality worldwide for 36 cancers in 185 countries[J]. CA-A Cancer Journal for Clinicians, 2018, 68(6): 394-424. DOI:10.3322/caac.21492 |
| [2] |
ZNAOR A, LORTET-TIEULENT J, LAVERSANNE M A, et al. International variations and trends in renal cell carcinoma incidence and mortality[J]. European Urology, 2015, 67(3): 519-530. DOI:10.1016/j.eururo.2014.10.002 |
| [3] |
SCELO G, LAROSE T L. Epidemiology and risk factors for kidney cancer[J]. Journal of Clinical Oncology, 2018, 36: 3574-3581. DOI:10.1200/JCO.2018.79.1905 |
| [4] |
LJUNGBERG B, BENSALAH K, CANFIELD S, et al. EAU guidelines on renal cell carcinoma:2014 update[J]. European Urology, 2015, 67(5): 913-924. DOI:10.1016/j.eururo.2015.01.005 |
| [5] |
GAUTIER L, COPE L, BOLSTAD B M, et al. Affy-analysis of affymetrix GeneChip data at the probe level[J]. Bioinformatics, 2004, 20(3): 307-315. DOI:10.1093/bioinformatics/btg405 |
| [6] |
RITCHIE M E, PHIPSON B, WU D, et al. Limma powers differential expression analyses for RNA-sequencing and microarray studies[J]. Nucleic Acids Research, 2015, 43(7): e47. DOI:10.1093/nar/gkv007 |
| [7] |
LANGFELDER P, HORVATH S. WGCNA:an R package for weighted correlation network analysis[J]. BMC Bioinforma-tics, 2008, 9: 559. DOI:10.1186/1471-2105-9-559 |
| [8] |
JEFFERY J, SINHA D, SRIHARI S, et al. Beyond cytokinesis:the emerging roles of CEP55 in tumorigenesis[J]. Oncogene, 2016, 35(6): 683-690. DOI:10.1038/onc.2015.128 |
| [9] |
ZHANG Weijing, NIU Chunhao, HE Weiling, et al. Upregulation of centrosomal protein 55 is associated with unfavorable prognosis and tumor invasion in epithelial ovarian carcinoma[J]. Tumor Biology, 2016, 37(5): 6239-6254. DOI:10.1007/s13277-015-4419-6 |
| [10] |
CHENG W Y, YANG T H, ANASTASSIOU D. Biomolecular events in cancer revealed by attractor metagenes[J]. PLoS Computational Biology, 2013, 9(2): e1002920. DOI:10.1371/journal.pcbi.1002920 |
| [11] |
FANG Yifeng, YU Hong, LIANG Xiao, et al. Chk1-induced CCNB1 overexpression promotes cell proliferation and tumor growth in human colorectal cancer[J]. Cancer Biology & Therapy, 2014, 15(9): 1268-1279. |
| [12] |
PFAFF K L, KING R W. Determinants of human cyclin B1 association with mitotic chromosomes[J]. PLoS One, 2013, 8(3): e59169. DOI:10.1371/journal.pone.0059169 |
| [13] |
WANG Z, SLIPICEVIC A, FORSUND M, et al. Expression of CDK1(Tyr15), pCDK1(Thr161), cyclin B1(total)and pCyclin B1(Ser126)in vulvar squamous cell carcinoma and their relations with clinicopatological features and prognosis[J]. PLoS One, 2015, 10(4): e0121398. DOI:10.1371/journal.pone.0121398 |
| [14] |
郭云韬, 喻超, 陈礼闻, 等. siRNA干扰沉默NUF2基因对肝癌HCCLM3细胞迁移和侵袭的影响[J]. 贵州医科大学学报, 2017, 42(2): 147-150. |
| [15] |
FU H L, SHAO L. Silencing of NUF2 inhibits proliferation of human osteosarcoma Saos-2 cells[J]. European Review for Medical and Pharmacological Sciences, 2016, 20(6): 1071-1079. |
| [16] |
HU Peng, SHANGGUAN Jianying, ZHANG Leida. Downregulation of NUF2 inhibits tumor growth and induces apoptosis by regulating lncRNA AF339813[J]. International Journal of Clinical and Experimental Pathology, 2015, 8(3): 2638-2648. |
| [17] |
MA Qing, LIU Yanmei, SHANG Liang, et al. The FOXM1/BUB1B signaling pathway is essential for the tumorigenicity and radioresistance of glioblastoma[J]. Oncology Reports, 2017, 38(6): 3367-3375. |
| [18] |
FU Xin, CHEN Guo, CAI Zhiduan, et al. Overexpression of BUB1B contributes to progression of prostate cancer and predicts poor outcome in patients with prostate cancer[J]. Onco Targets and Therapy, 2016, 9(Issue 1): 2211-2220. |
| [19] |
ZHANG Yixiang, YUAN Yeqing, LIANG Pei, et al. Overexpression of a novel candidate oncogene KIF14 correlates with tumor progression and poor prognosis in prostate cancer[J]. Oncotarget, 2017, 8(28): 45459-45469. |
| [20] |
LI K K, QI Y, XIA T, et al. The kinesin KIF14 is overexpressed in medulloblastoma and downregulation of KIF14 suppressed tumor proliferation and induced apoptosis[J]. Laboratory Investigation, 2017, 97(8): 946-961. DOI:10.1038/labinvest.2017.48 |
| [21] |
QIU H L, DENG S Z, LI C, et al. High expression of KIF14 is associated with poor prognosis in patients with epithelial ovarian cancer[J]. European Review for Medical and Pharmacological Sciences, 2017, 21(2): 239-245. |
| [22] |
PAN Xiuwu, CHEN Lu, HONG Yi, et al. EIF3D silencing suppresses renal cell carcinoma tumorigenesis via inducing G2/M arrest through downregulation of Cyclin B1/CDK1 signaling[J]. International Journal of Oncology, 2016, 48(6): 2580-2590. DOI:10.3892/ijo.2016.3459 |
| [23] |
XU Leilei, XIA Chao, SHENG Fei, et al. CEP55 promotes the proliferation and invasion of tumour cells via the AKT signalling pathway in osteosarcoma[J]. Carcinogenesis, 2018, 39(4): 623-631. DOI:10.1093/carcin/bgy017 |
| [24] |
ZHAO W M, SEKI A, FANG G W. Cep55, a microtubule-bundling protein, associates with centralspindlin to control the midbody integrity and cell abscission during cytokinesis[J]. Molecular Biology of the Cell, 2006, 17(9): 3881-3896. DOI:10.1091/mbc.e06-01-0015 |
| [25] |
LIU Qiang, DAI Shejiao, LI Hong, et al. Silencing of NUF2 inhibits tumor growth and induces apoptosis in human hepatocellular carcinomas[J]. Asian Pacific Journal of Cancer Prevention, 2014, 15(20): 8623-8629. DOI:10.7314/APJCP.2014.15.20.8623 |
| [26] |
YANG Ye, GU Chunyan, LUO Chen, et al. BUB1B promotes multiple myeloma cell proliferation through CDC20/CCNB axis[J]. Medical Oncology, 2015, 32(3): 81. DOI:10.1007/s12032-015-0542-x |
| [27] |
XU H, CHOE C, SHIN S H, et al. Silencing of KIF14 interferes with cell cycle progression and cytokinesis by blocking the p27Kip1 ubiquitination pathway in hepatocellular carcinoma[J]. Experimental & Molecular Medicinee, 2014, 46(5): e97. |
| [28] |
HUANG S K, QIAN J X, YUAN B Q, et al. SiRNA-mediated knockdown against NUF2 suppresses tumor growth and induces cell apoptosis in human glioma cells[J]. Cellular and Molecular Biology, 2014, 60(4): 30-36. |
 2019, Vol. 55
2019, Vol. 55

